超详细的保姆级SnapGene软件应用汇总,建议收藏备用!!!
- 农业机械
- 2025-03-04 17:49:06
- 114

同时小编在这里说明,支持正版SnapGene软件(懂的都懂)!!!这期内容讲解主要应用版本进行实战讲解。下面是本期课程的内容框架,基本大致围绕框架进行讲解,但会着重介绍软件功能应用!!!本期内容框架1......

同时小编在这里说明,支持正版SnapGene软件(懂的都懂)!!!这期内容讲解主要应用版本进行实战讲解。下面是本期课程的内容框架,基本大致围绕框架进行讲解,但会着重介绍软件功能应用!!!

本期内容框架
1
SnapGene软件概览
1.1SnapGene软件简介
SnapGene软件是一款强大的基因编辑软件,它可以帮助科研小伙伴们更轻松地进行基因组编辑和研究。同时,SnapGene软件支持多种基因编辑工具,包括CRISPR/Cas9、TALEN、ZFN等,还包含了序列编辑和标记、质粒图谱构建、酶切位点分析、序列比对等多种功能,可以满足不同生物学研究人员对相应序列进行分析的操作。
不仅如此,还是一款强大而易于使用的分子生物学实验设计和分析工具,帮助科学家和研究人员更高效地进行基因工程和分子生物学研究。它提供了直观的界面、丰富的功能和可视化工具,简化了实验设计、数据分析和结果展示的过程。
1.2SnapGene软件用途
1.2.1DNA序列编辑和设计
SnapGene提供了直观的界面,可用于编辑、设计和构建DNA序列。用户可以进行序列插入、删除、复制和反转等操作,以及添加或修改限制性内切酶位点。此外,SnapGene还支持PCR引物设计、引物扩增和序列组装等功能。
1.2.2限制酶酶切图谱分析
SnapGene可以自动分析DNA序列中的限制酶切位点,并生成直观的酶切图谱。用户可以查看DNA片段的预测酶切模式,快速评估酶切方案的可行性,并选择合适的酶切酶进行实验。
1.2.3转录和翻译分析
SnapGene可以帮助用户分析DNA序列的转录和翻译过程。它提供了转录起始位点和终止位点的预测,并可显示编码蛋白质的氨基酸序列。用户可以直观地查看ORF(开放阅读框)和编码蛋白质的特性,以及序列中可能存在的转录调控元件。
1.2.4Plasmid图设计和构建
SnapGene支持Plasmid(质粒)图的设计、编辑和可视化。用户可以快速构建质粒图,添加、删除或修改基因、启动子、标签和其他功能元件。它还提供了可定制的注释和图形标记,使得质粒图更加清晰和易于理解。
1.2.5DNA文档和分享
SnapGene允许用户创建详细的DNA文档,包括序列信息、注释、图形标记和参考文献等。这些文档可以方便地保存、共享和印刷,以支持实验设计、报告撰写和实验室交流。
1.3版本安装
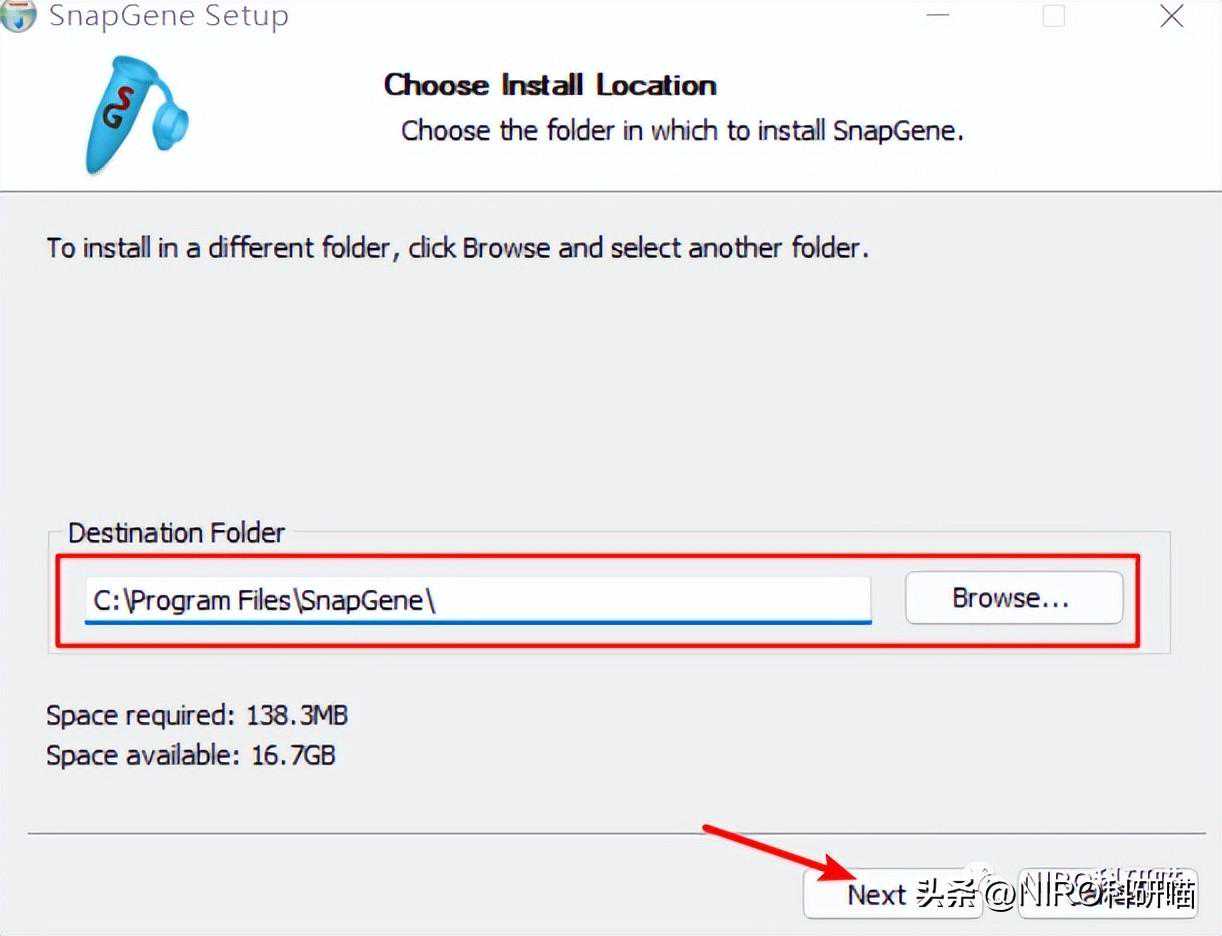
1.3.2默认路径,然后按顺序安装,直到安装完成即可。
2
怎么白嫖最新版SnapGene软件?
SnapGene软件几乎每年都会更新版本。更新版本后,有些新功能是旧版不能用的,例如6.0.2版本就是不能使用“goldengate”这一功能!!!那么问题来了,我们想使用一下“goldengate”这个功能,但是又不经常用,这时候我们该怎么办呢?每当这时候小编都会说一句“只要思想不滑坡,办法总比困难多”。小编在这里给大家介绍这么一个办法!!!
首先去SnapGene官网申请最新版本免费试用版!!!就可以免费白嫖1个月啦!!!只要不花钱,小编就觉得“香”!!!既省时又省力,那可真是太棒了!!!同学们,给我“香”起来,哈哈哈哈!开玩笑!!!说说它们的优缺点吧!!!
优点
毋庸置疑的就是我们可以使用到最新版本的功能,同时还可以白嫖最新版本使用1个月!!!不仅可以解决了眼前难题,又可以白嫖我们不常用的功能,真是省钱又省力的好软件啊!这也太香了吧!!!
缺点就是要先把我们现有的SnapGene软件卸载,重装最新版本SnapGene软件,为什么呢?首先是避免新旧软件覆盖的问题,出现路径不同,而导致报错;其次就是避免旧版本出现升级,然后不能使用旧版本,而要重新购买等问题。下面小编为大家介绍一下怎么申请SnapGene软件免费试用版吧!!!
首先,进入SnapGene官网()。
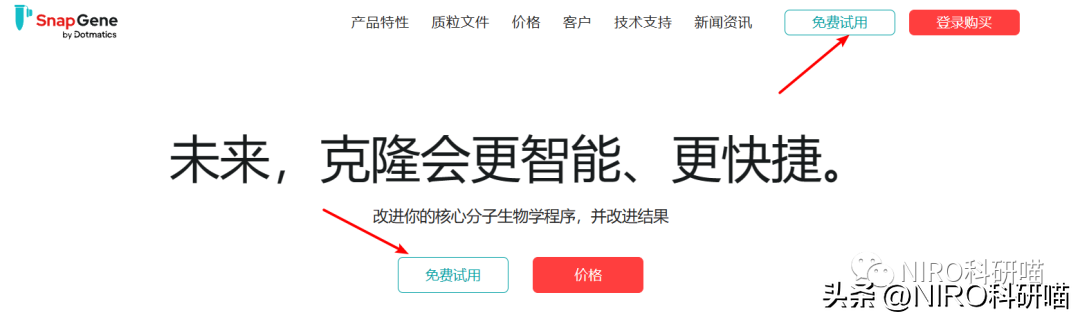
其次,输入邮箱,提交申请。
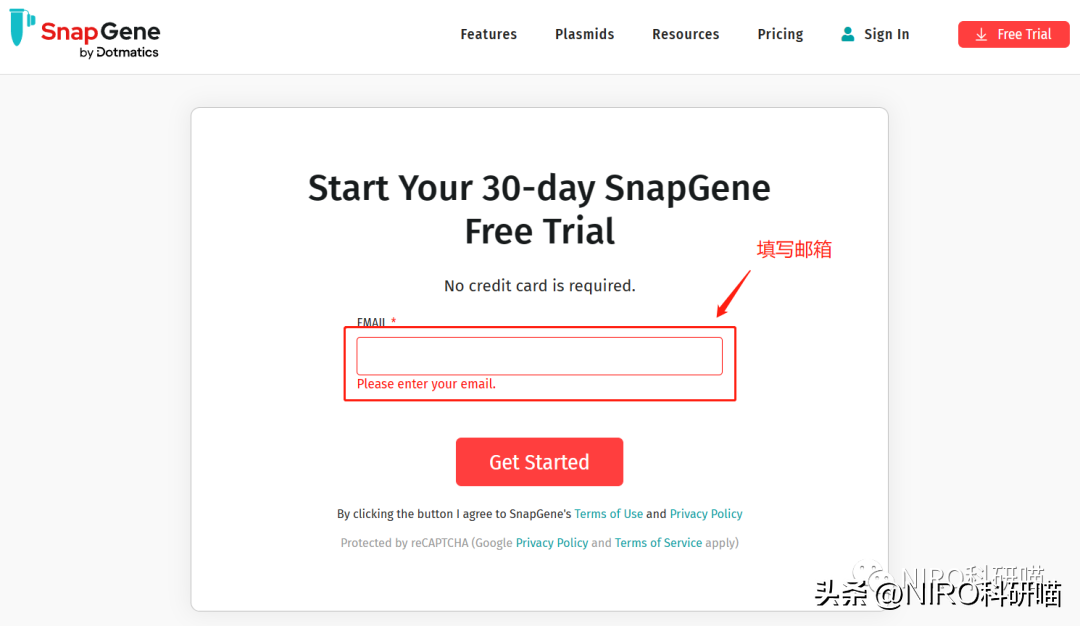
然后,按照下面的选择“Yes”和“Student”,提交。
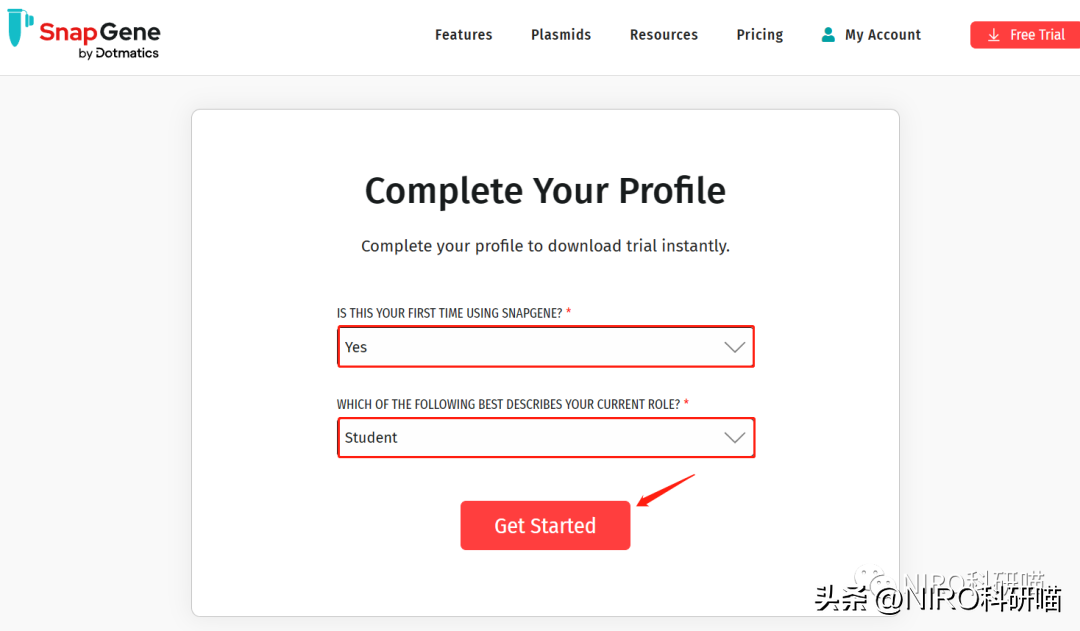
再然后,获取登录安装信息:组名、登记密码(激活软件)、试用截止日期。

最后,根据电脑系统,选择下载安装包,按顺序安装,输入组名、登录密码即可使用。
小编觉得版本和旧版本区别挺大的!!!首先是全新的欢迎界面,很大程度上提高了用户的体验感;其次是选项卡式界面、文件和文件夹管理文件及文件搜索,一定程度上提高我们的工作效率;最后是引物注释和新序列的预览检测功能,不仅添加/编辑引物注释,还增加新序列预览窗口。
3
SnapGene基本功能
3.1SnapGene软件基本界面(下面以6.0.2版本为例)
3.1.1版本的欢迎界面。
3.1.2版本左下角是可以转换语言的,有“中文”、“英语”、“日语”三种语言,小编在这里为了让大家更好更快熟悉界面,选择了中文语言。
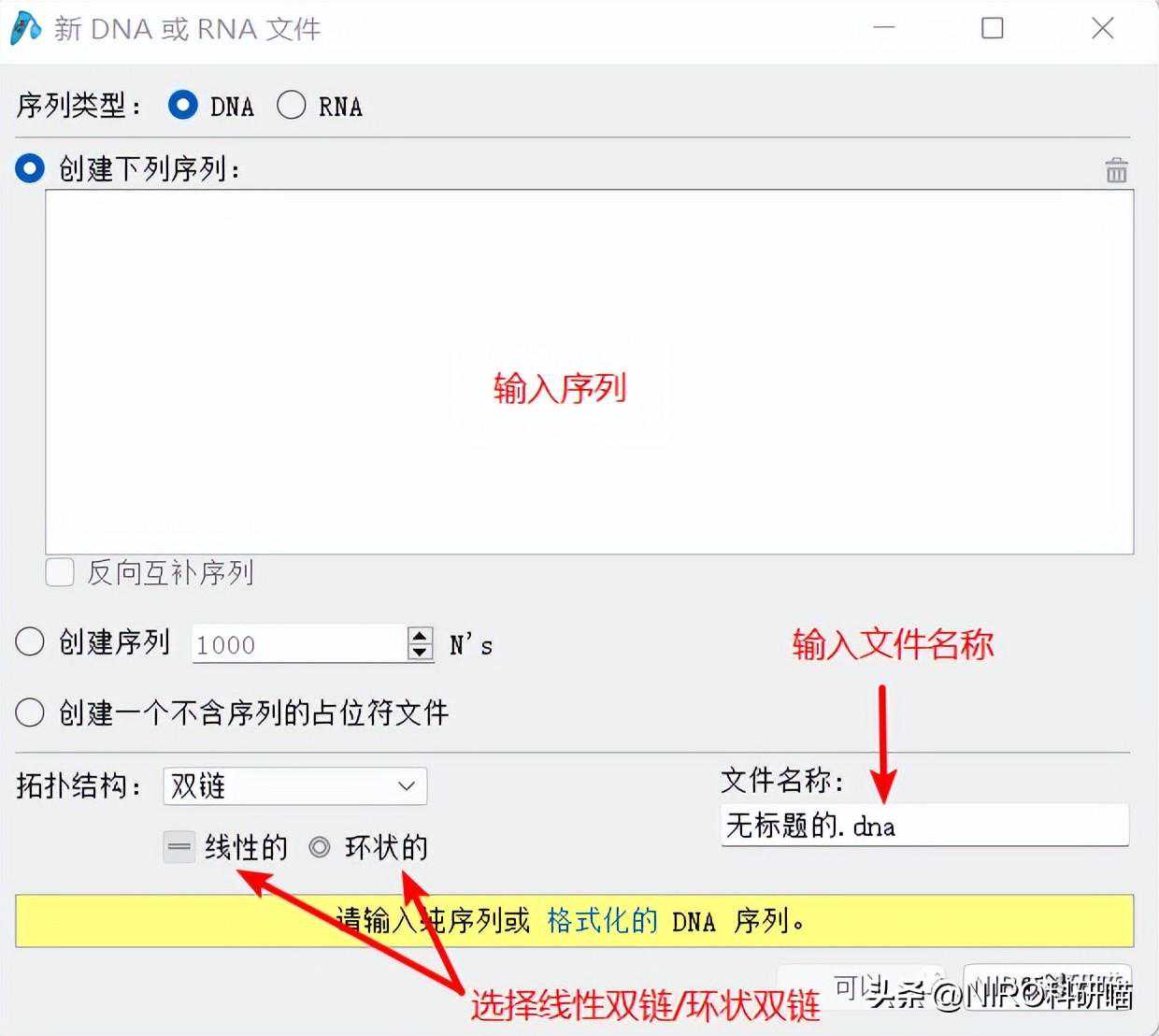
3.1.3版本新建DNA/RNA文件。
3.1.4打开文件。
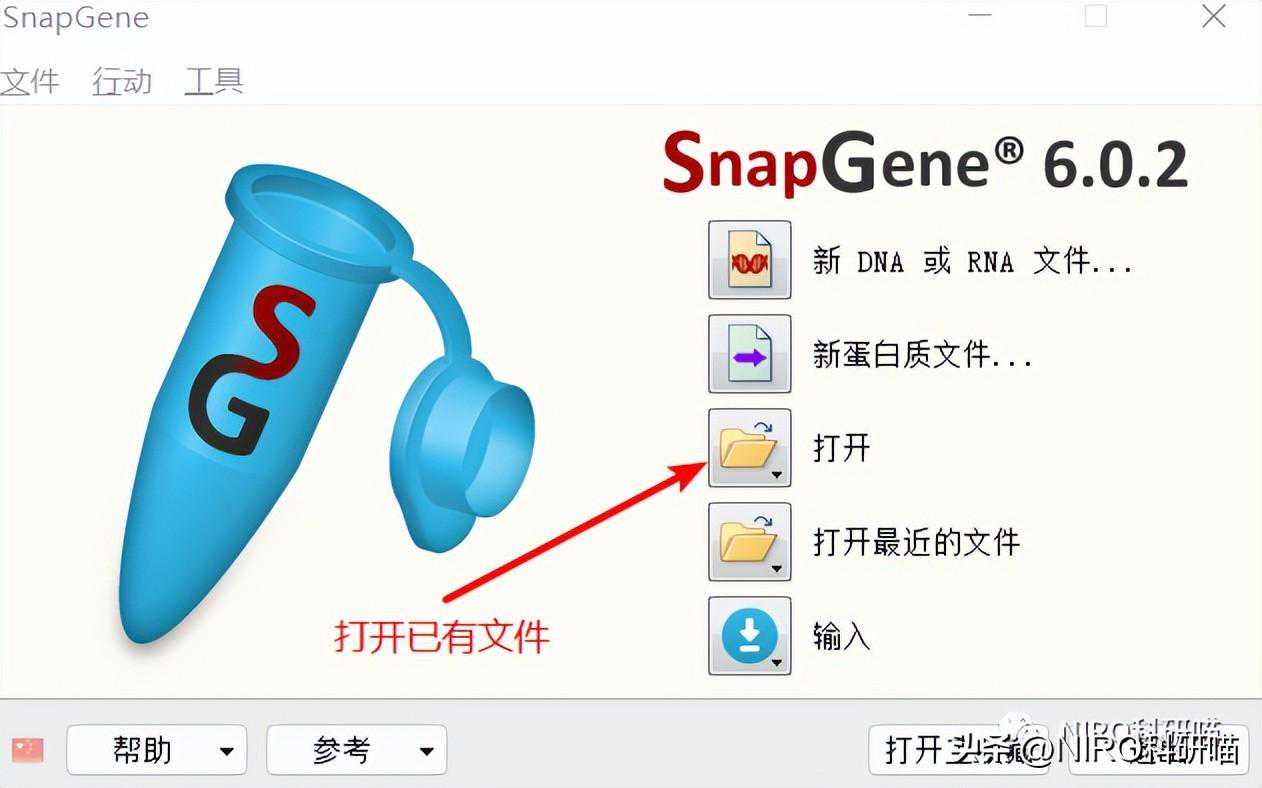
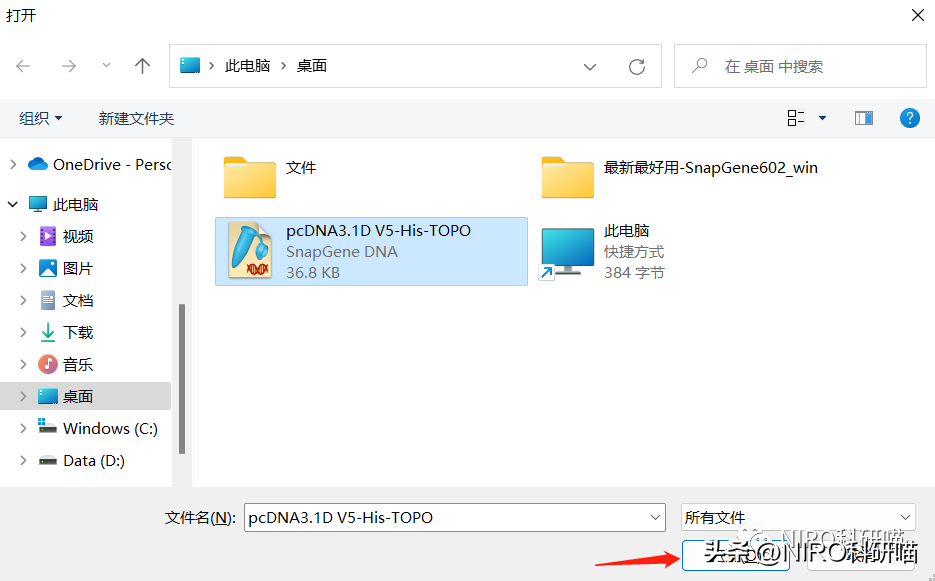
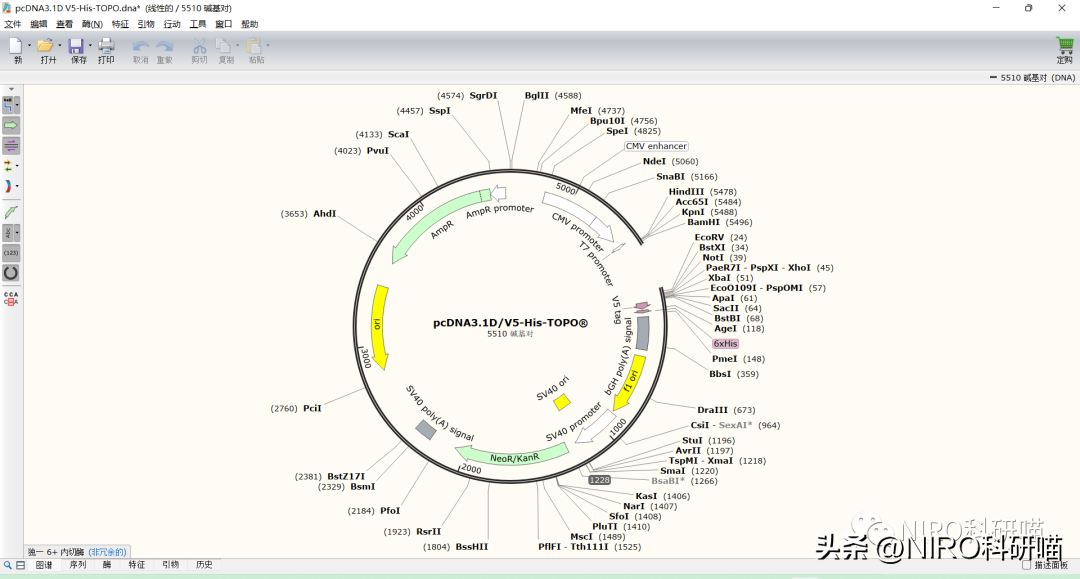
SnapGene官网()中有很多常见的质粒文件,如果找不到合适的可以到SnapGene官网中查询!!!
3.1.5导入文件。
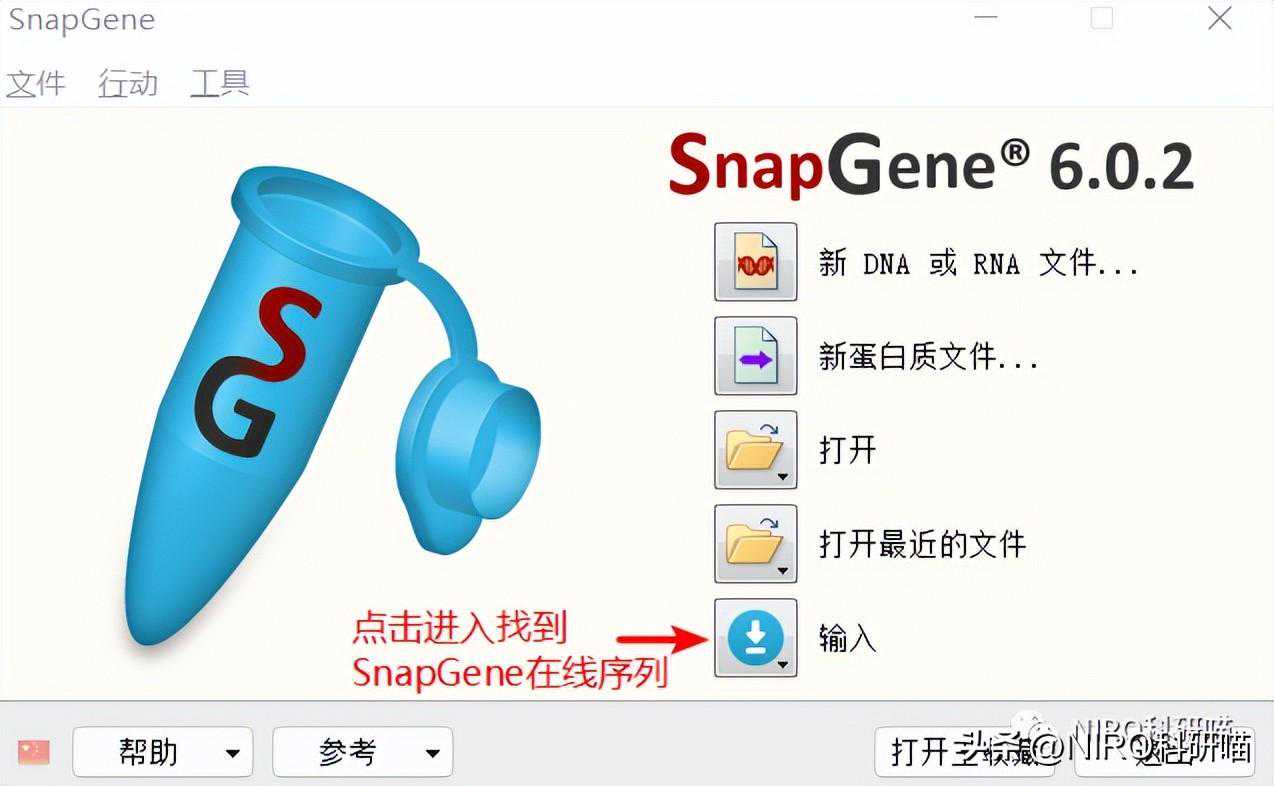
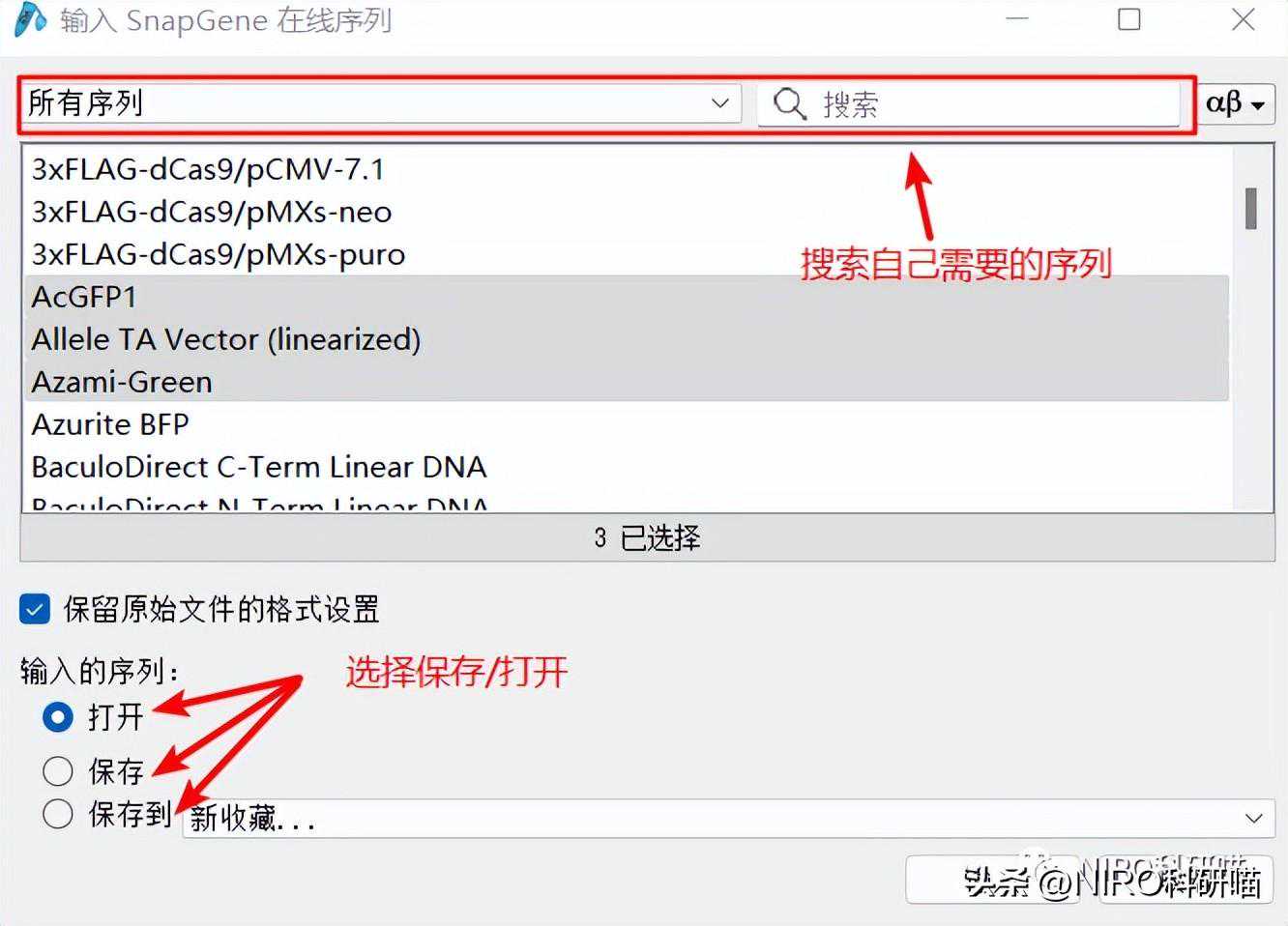
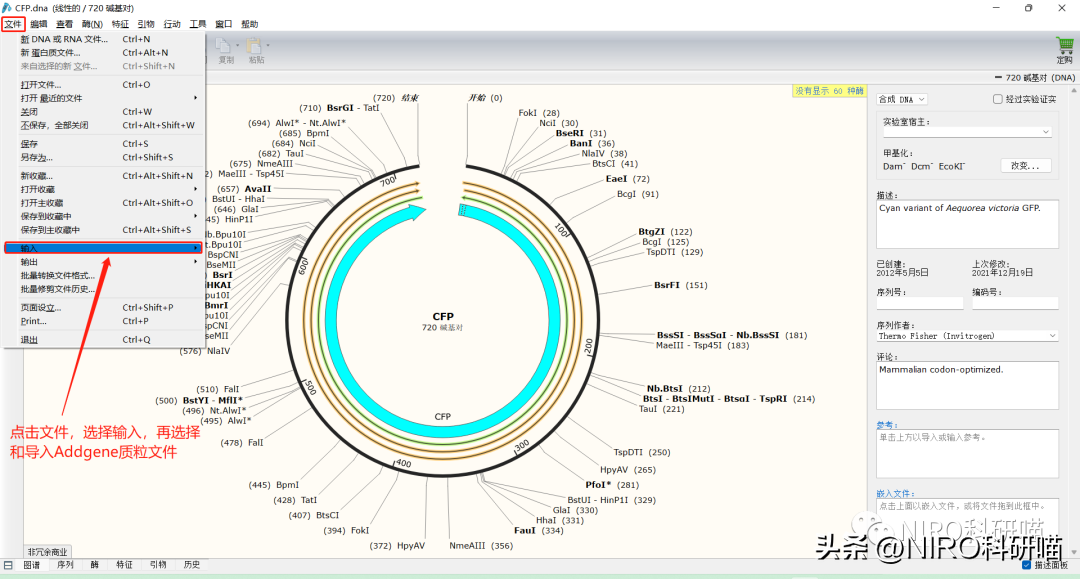
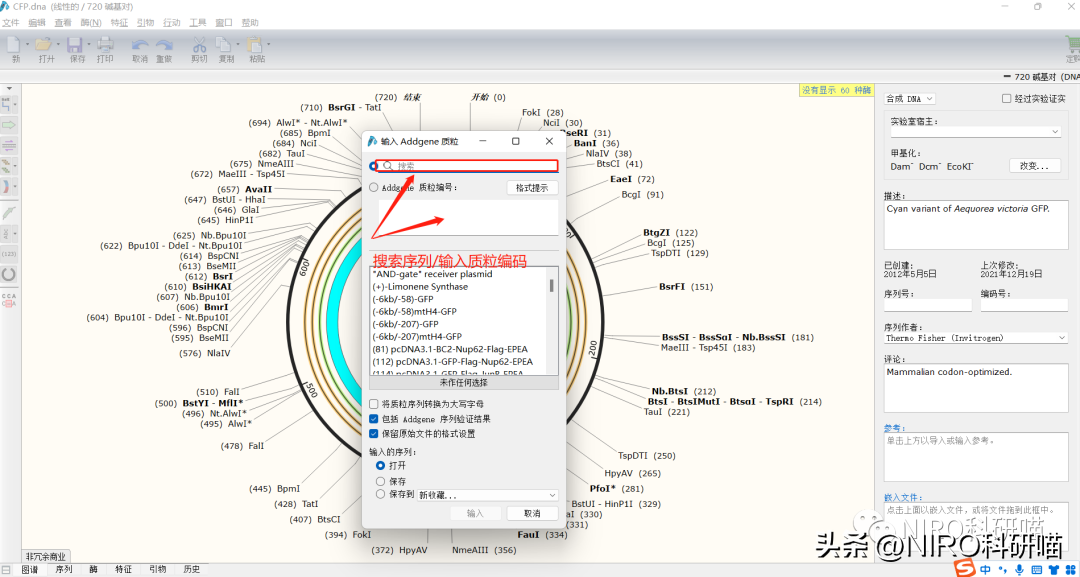
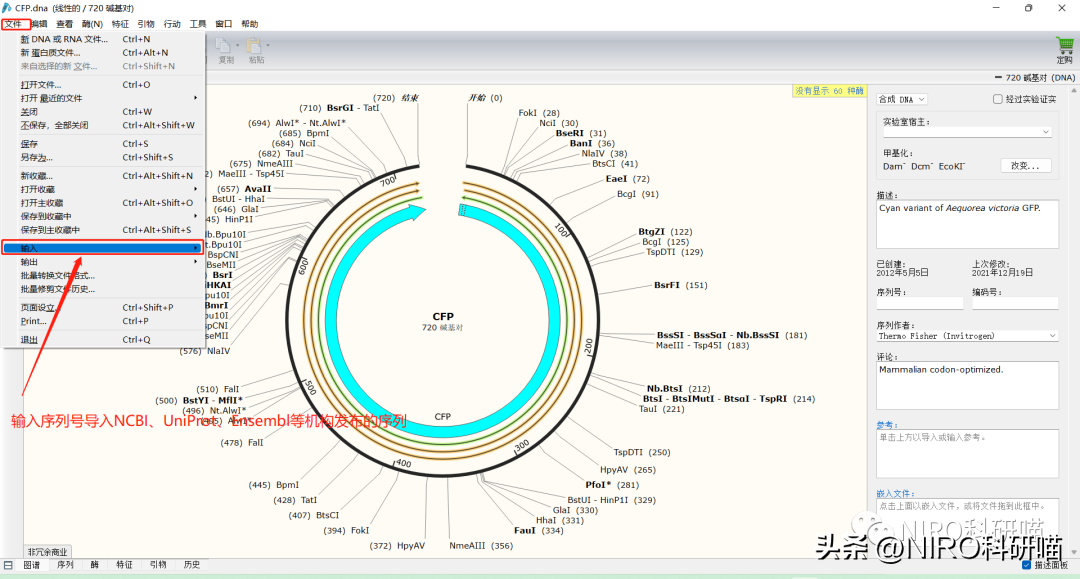
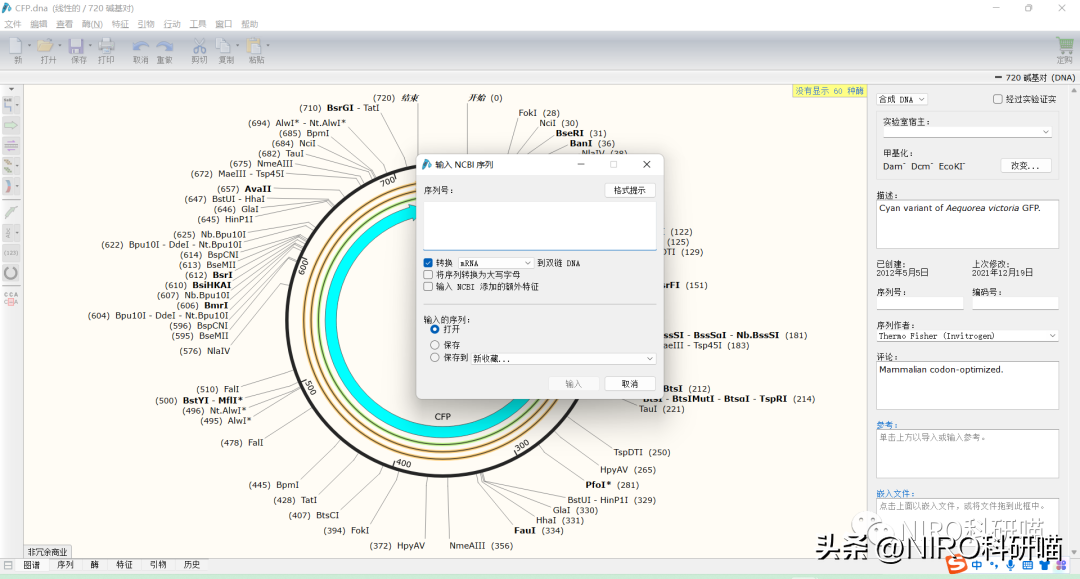
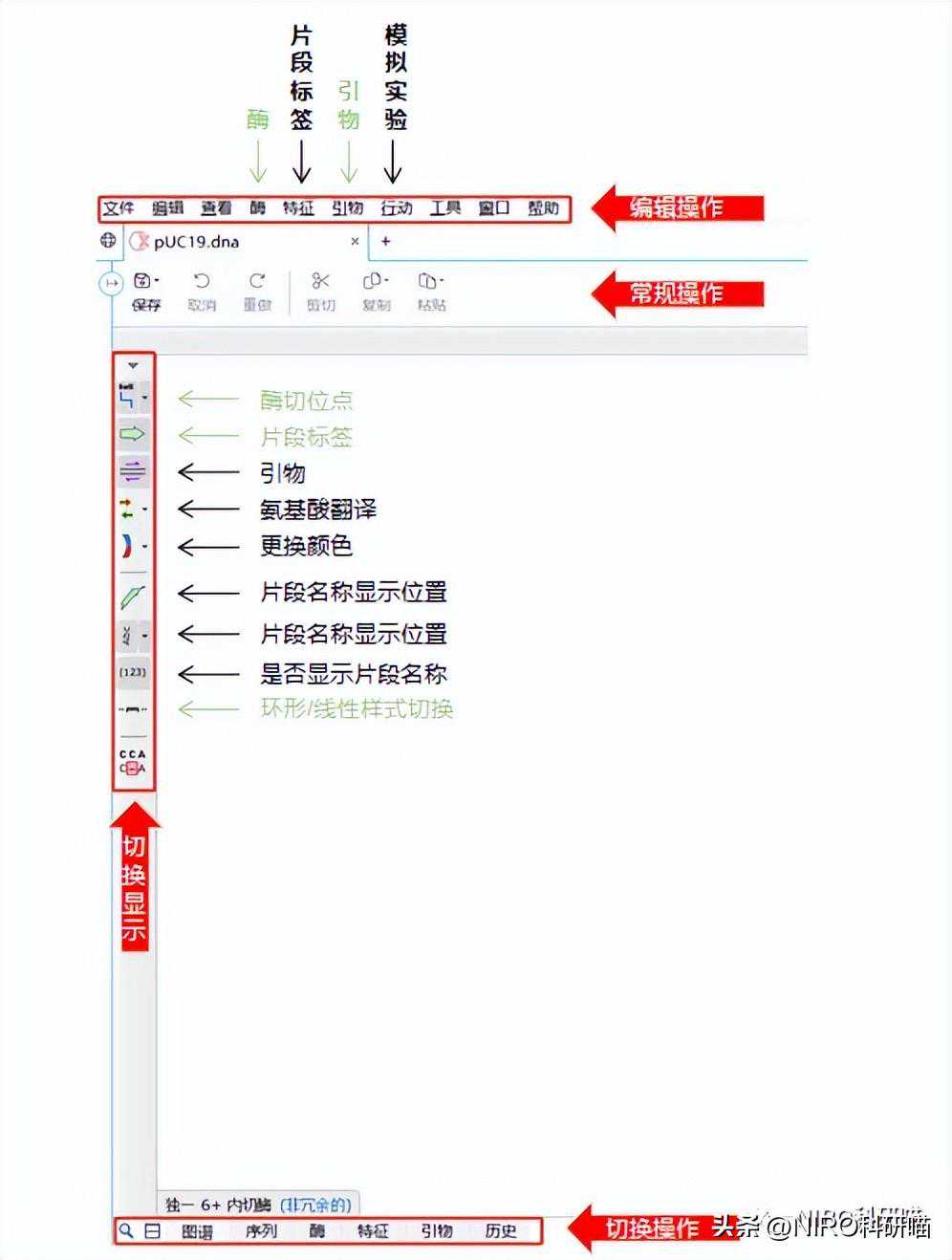
3.2工具栏介绍
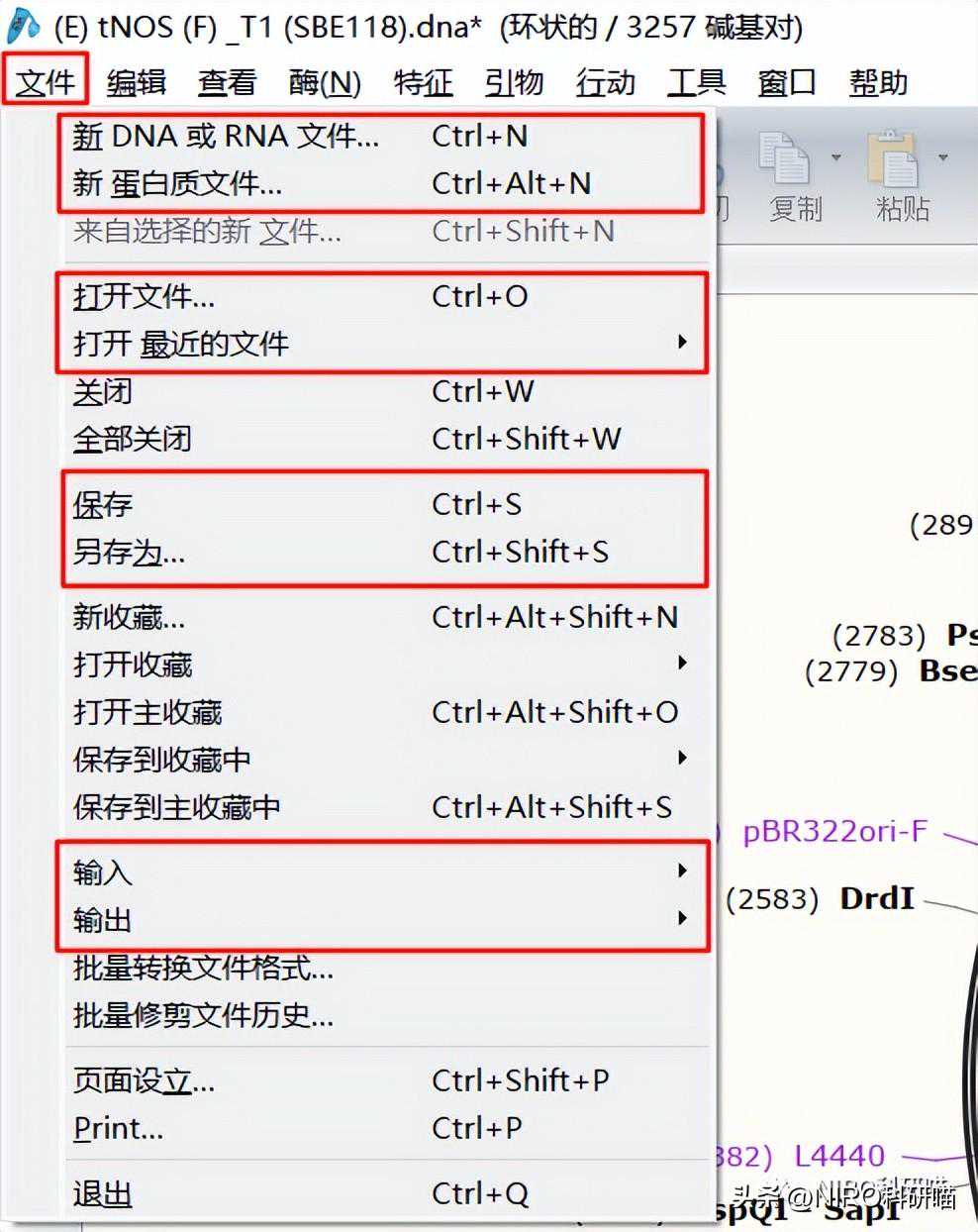
3.2.1.1文件
在文件中,标红框是经常使用的功能操作键。
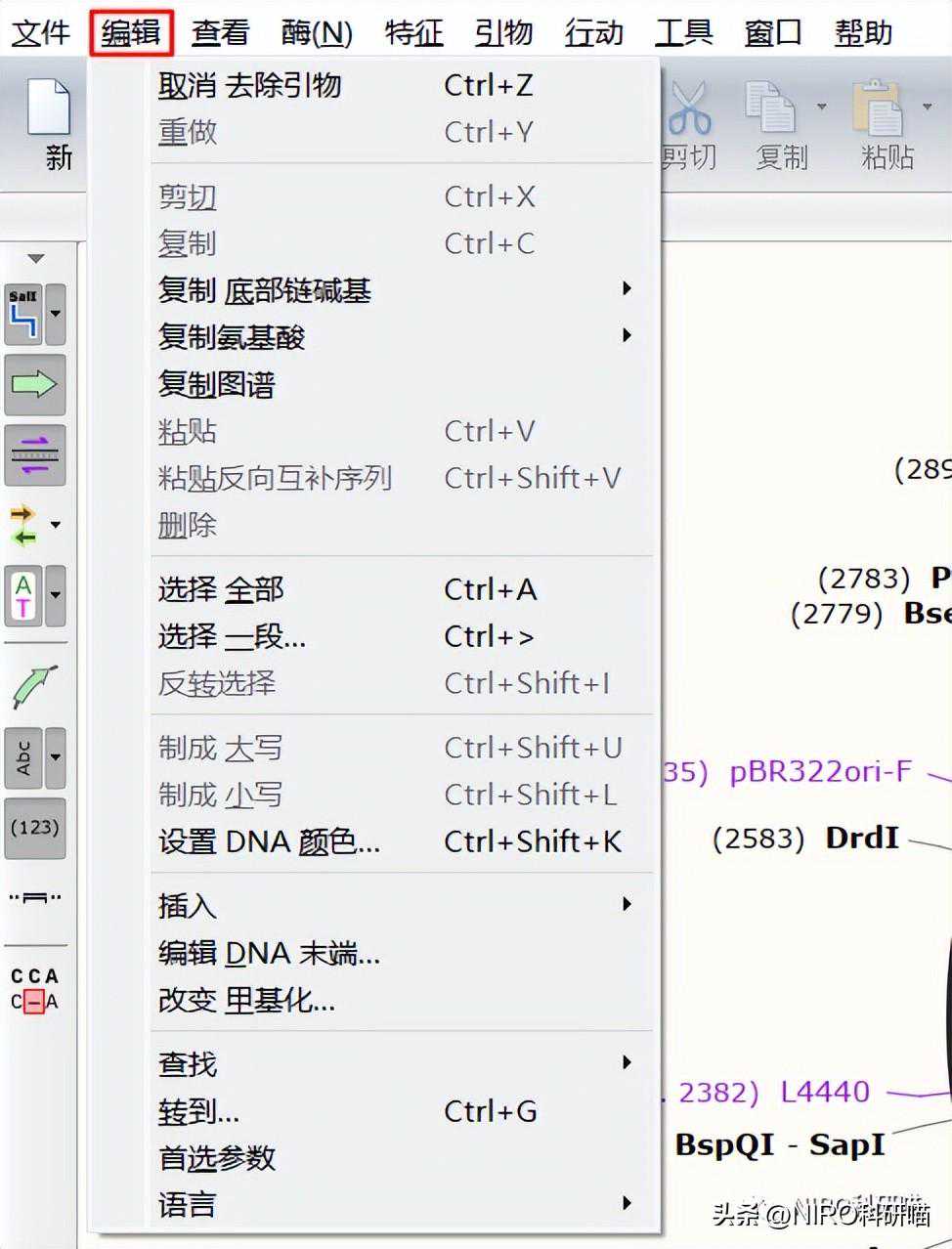
3.2.1.2编辑
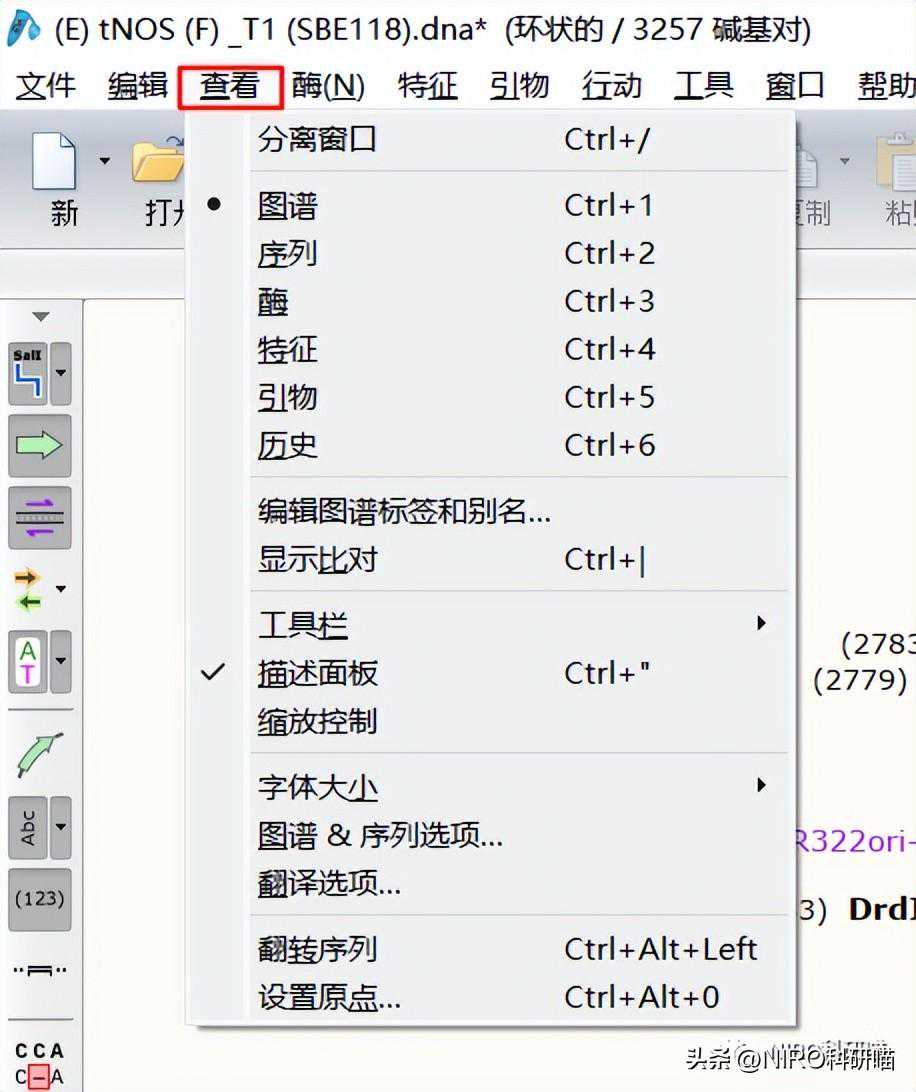
3.2.1.3查看
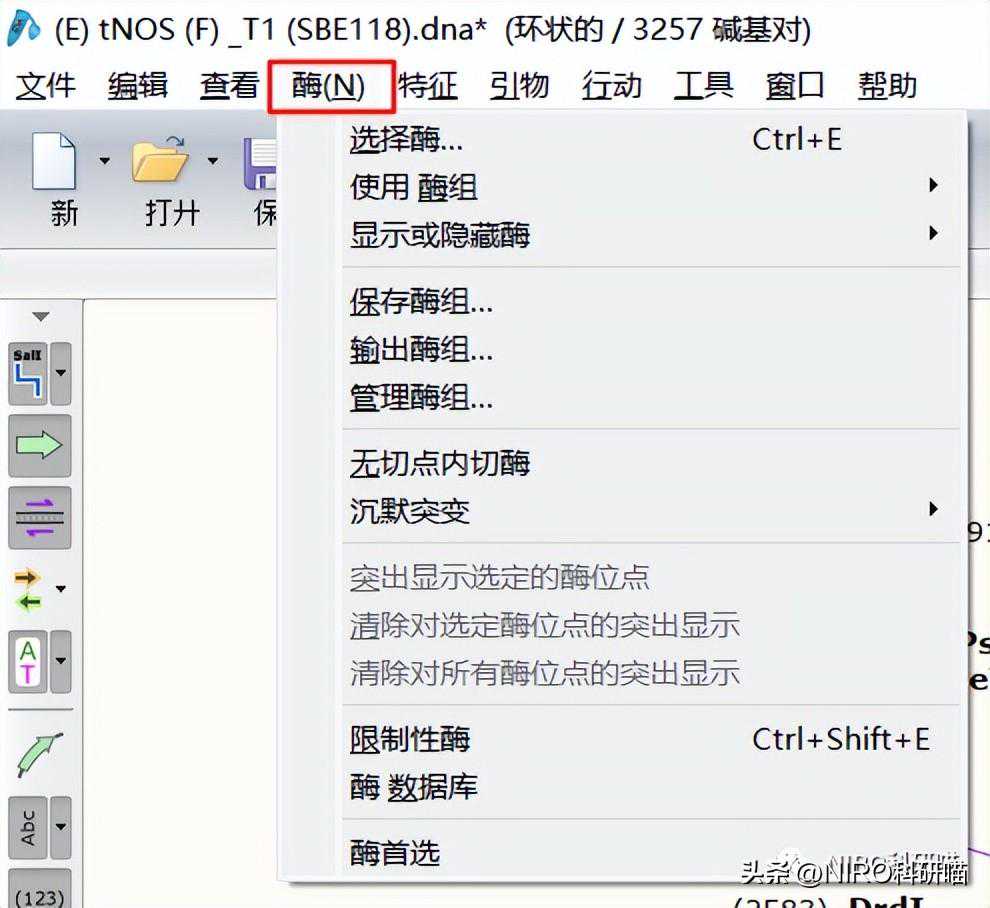
3.2.1.4查看
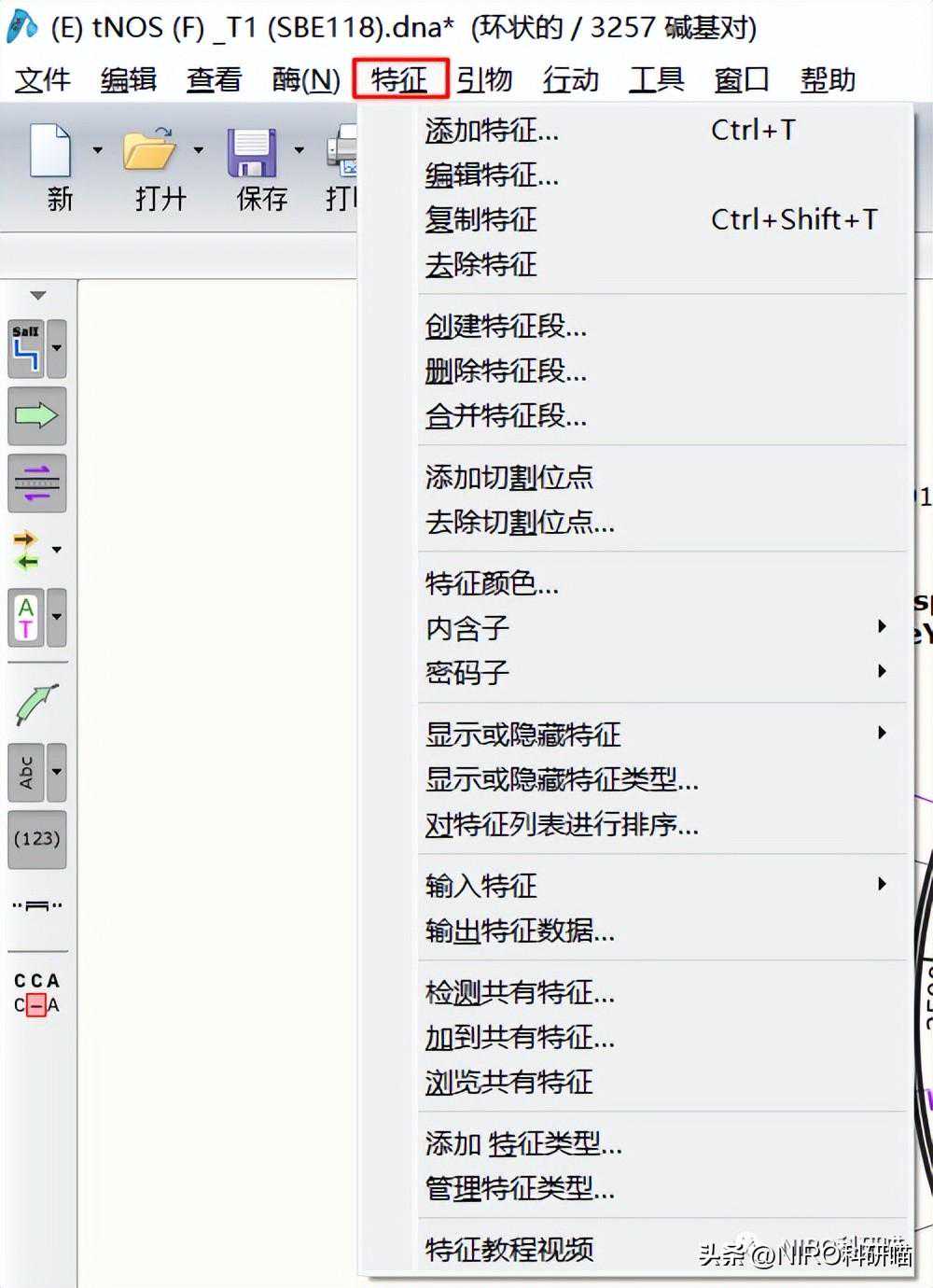
3.2.1.5特征
3.2.1.6引物
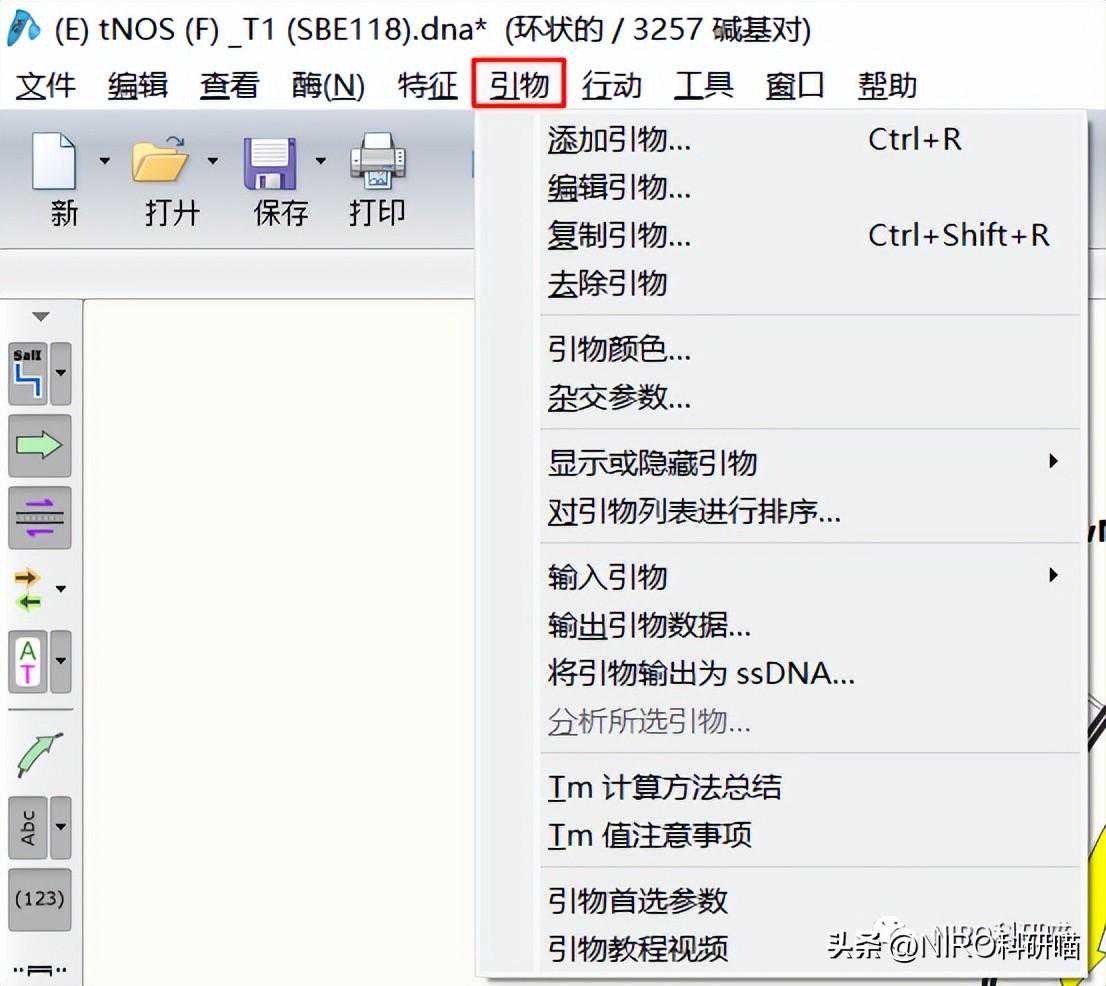
3.2.1.7行动
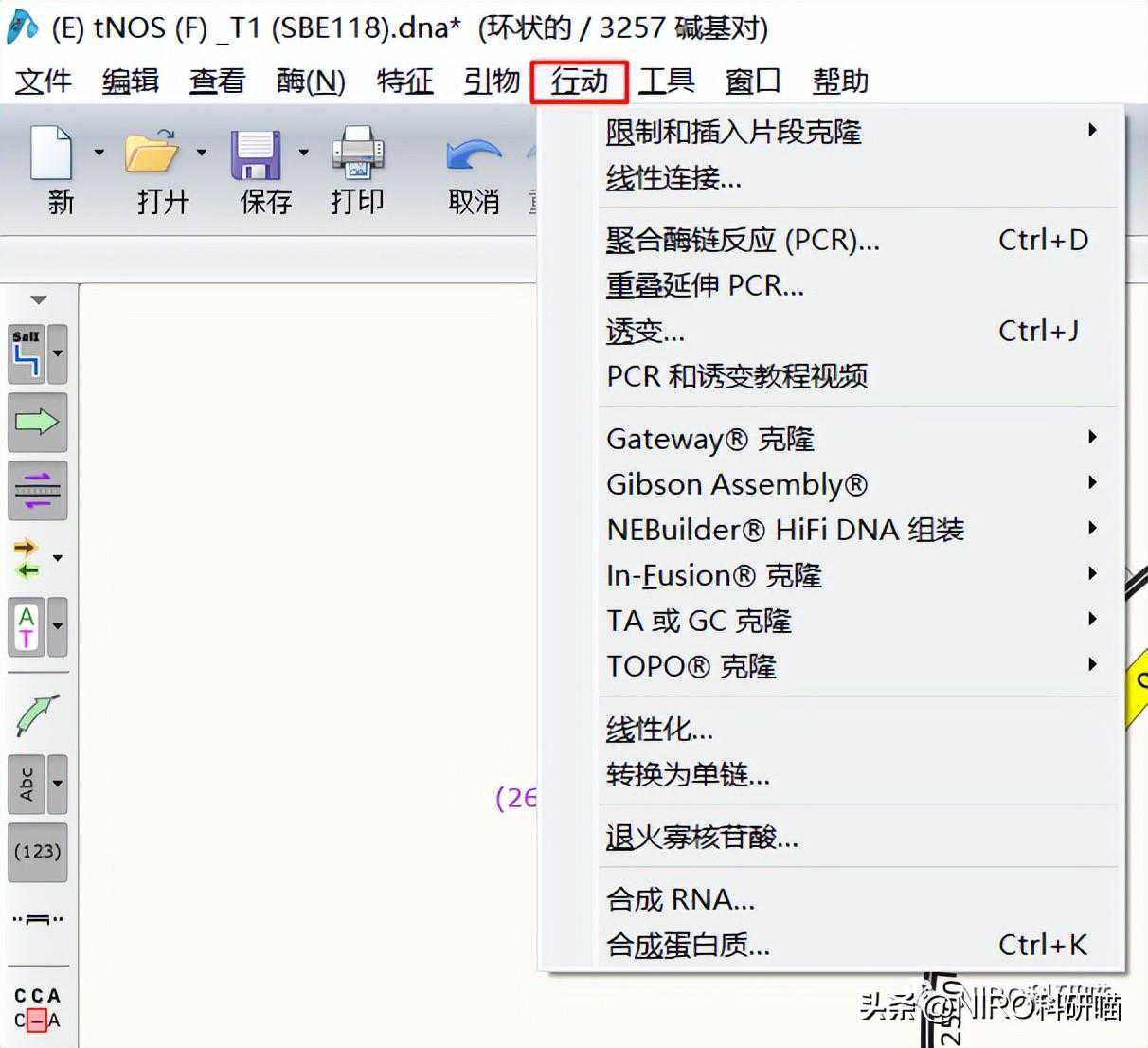
3.2.1.8工具
3.2.1.9窗口
3.2.1.10帮助
3.2.2左侧工具栏(编辑操作)
3.2.3底部工具栏(编辑操作)
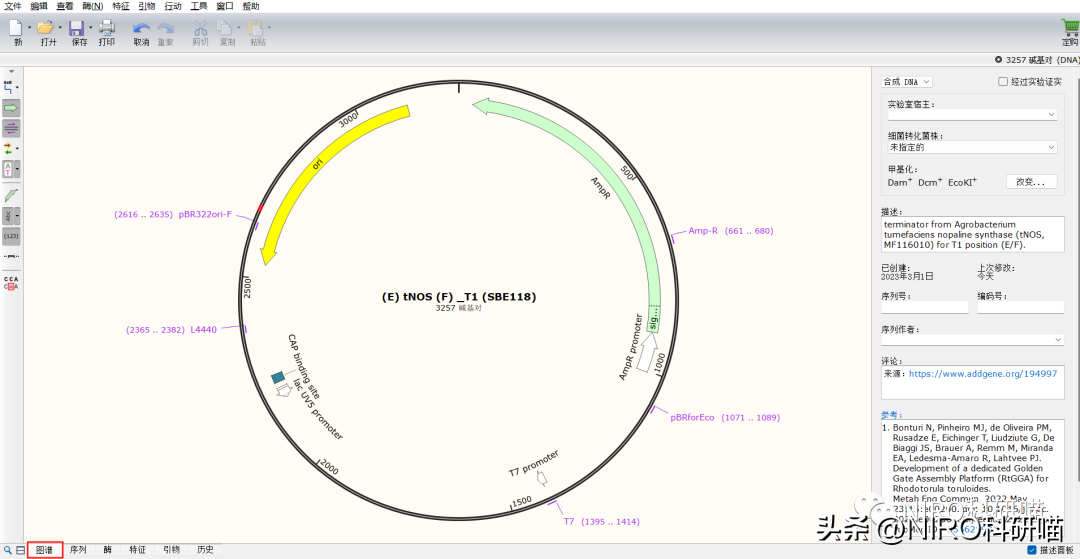
3.3.2.1图谱
3.2.2.2序列

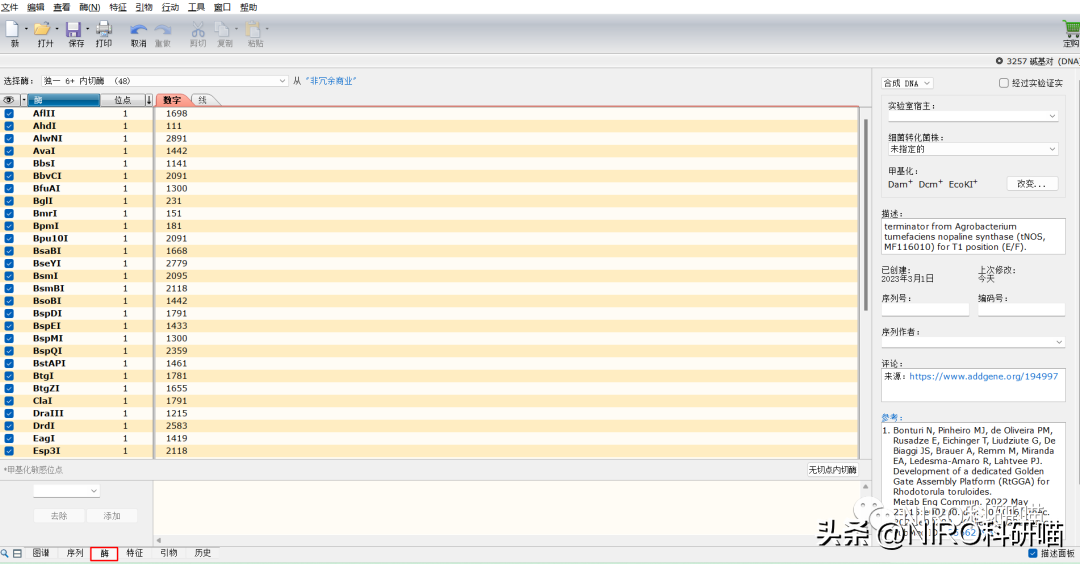
3.2.2.3酶
3.2.2.4特征
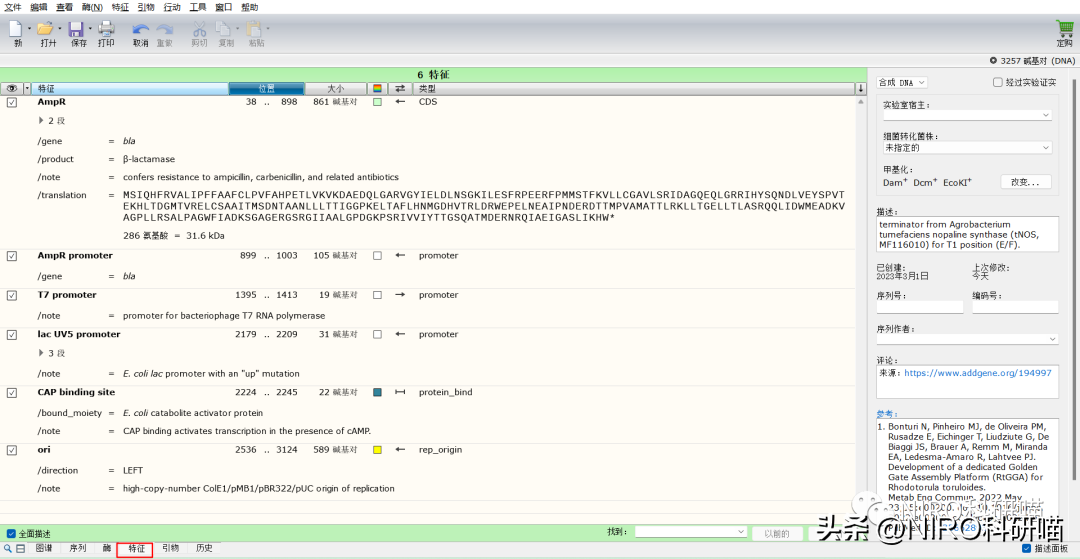
3.2.2.5引物
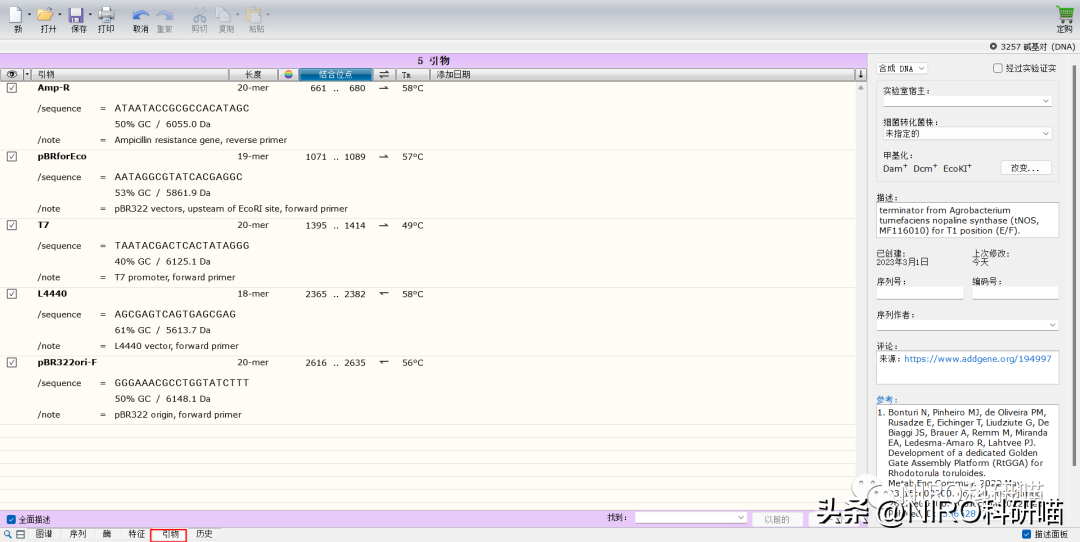
4
SnapGene软件应用
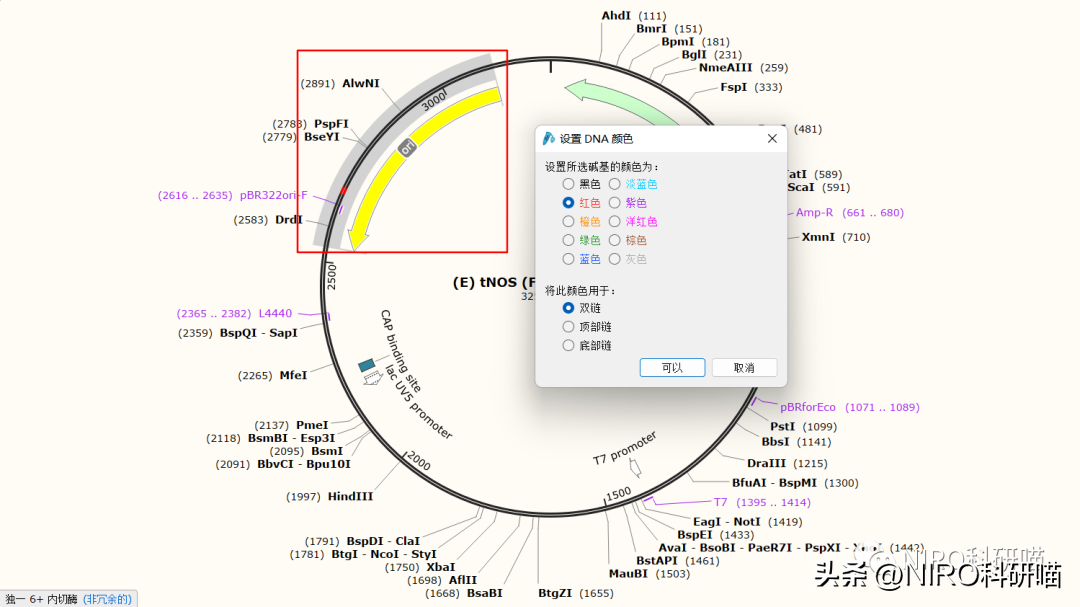
4.1自定义DNA序列配色
4.1.1选择一段序列区域

4.1.2自定义颜色序列
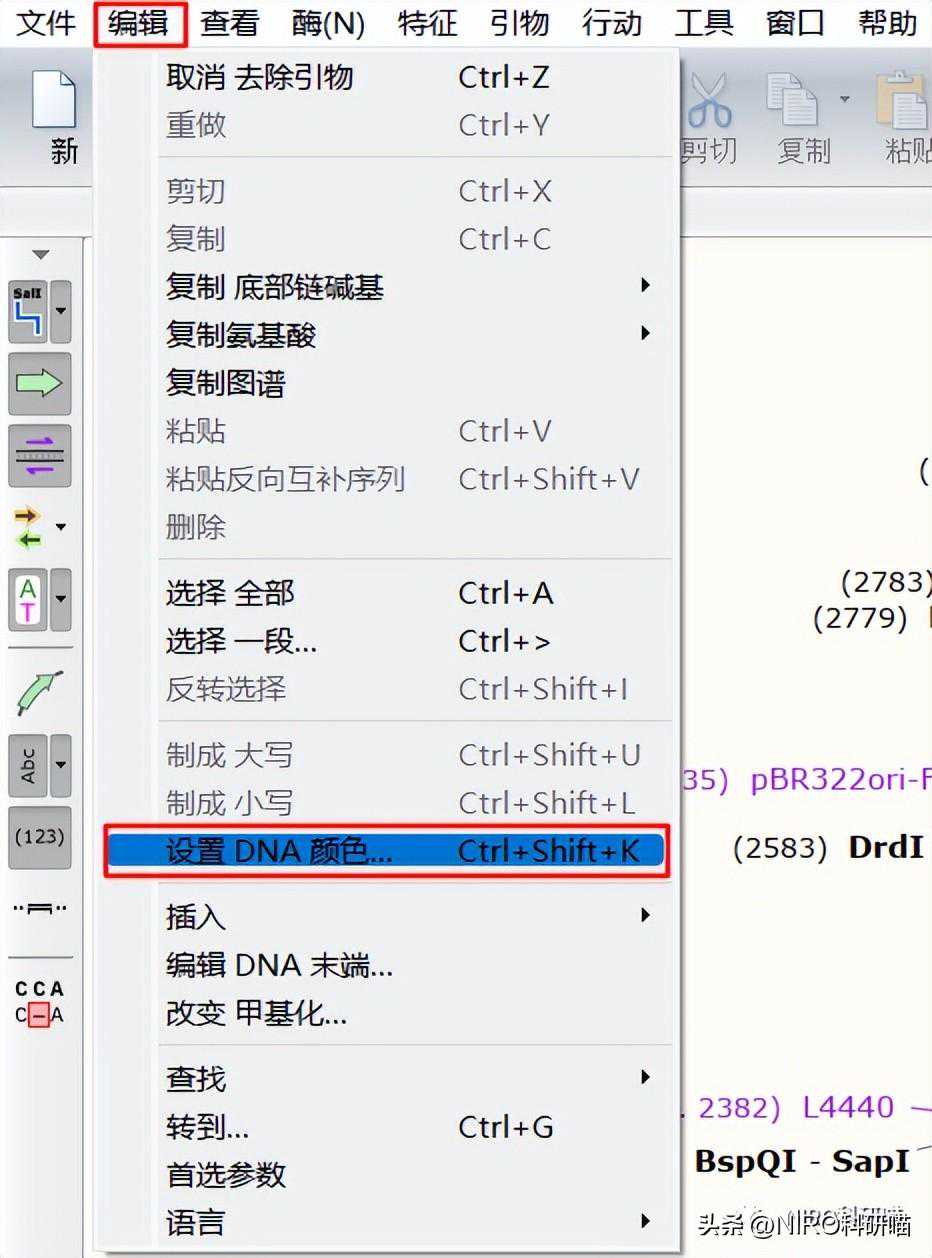
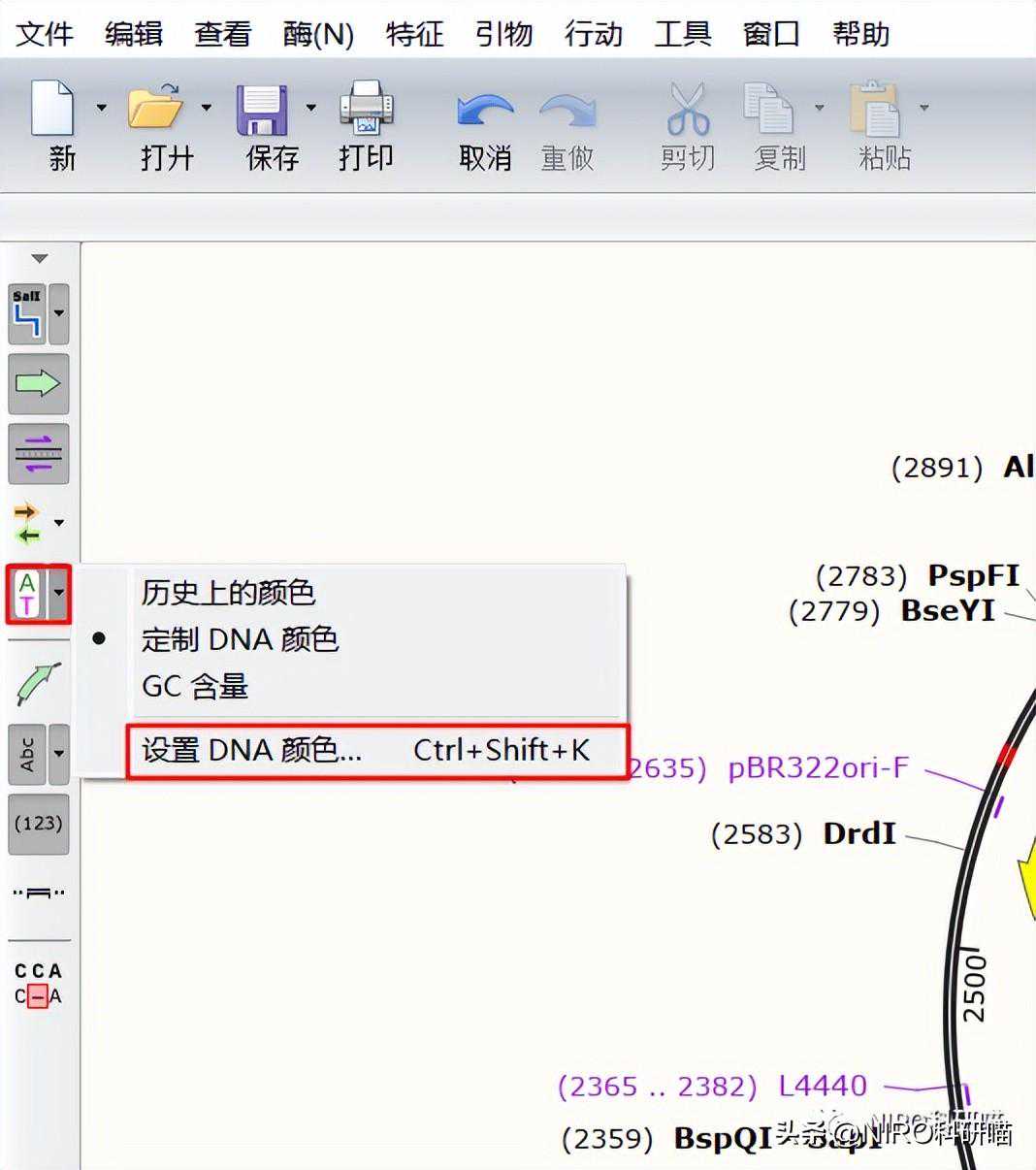
4.1.3选择配色
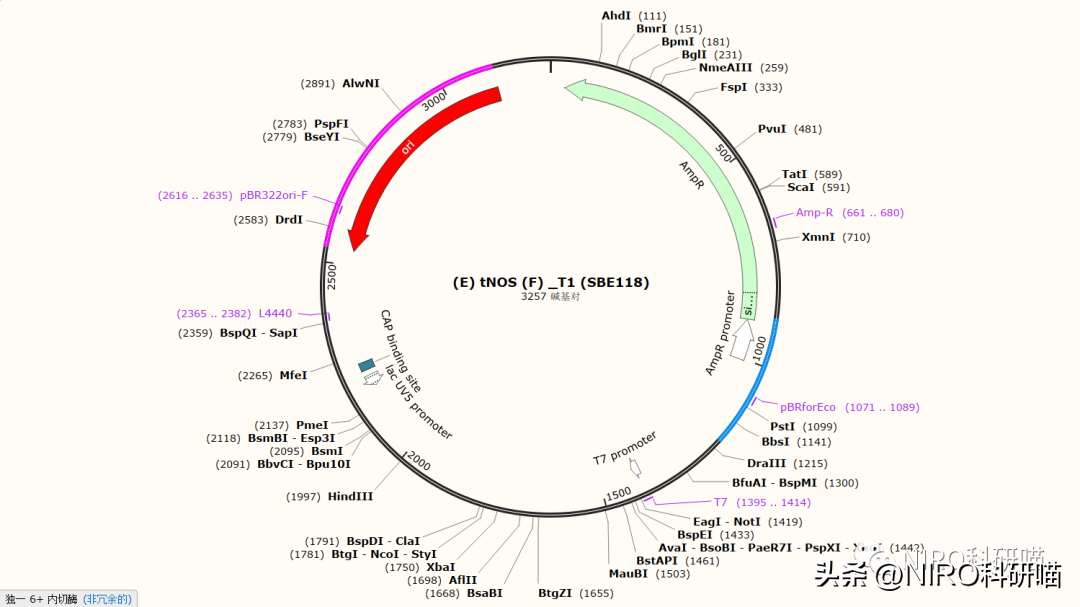
4.1.4预览自定义配色
4.2修改字体大小
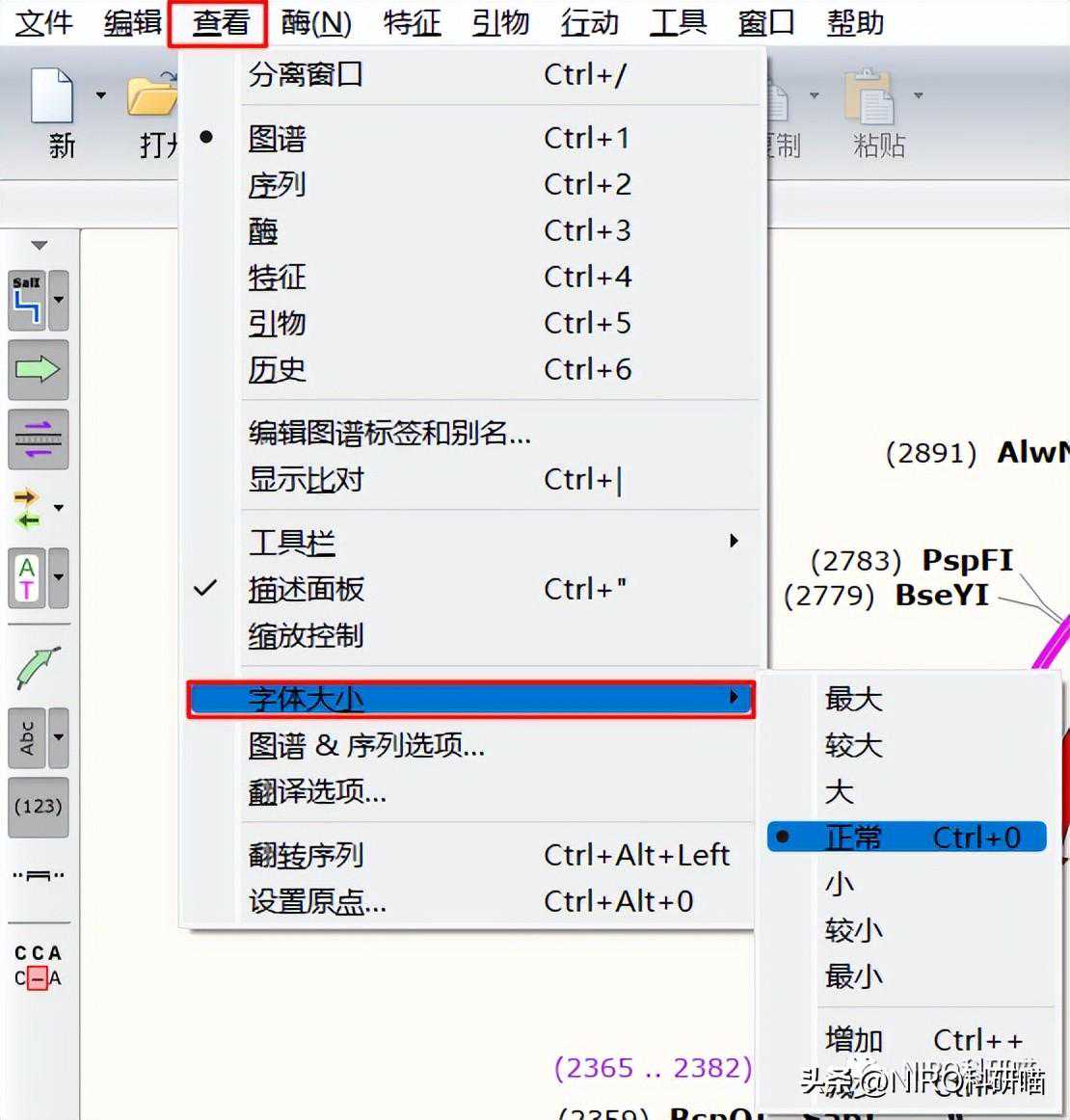
注:字号大小的修改会应用到SnapGene所有的文件,比如Map、序列、酶、引物、以及历史记录;字号大小会影响一些展示的宽度;如果SnapGene里的字号修改依然无法适配您的要求,请检查您电脑显示器分辨率的设置。
4.3导出为矢量图片
4.4获取载体序列
很多实验室同学,会经常遇到这样的情况,不知道从哪里获取载体序列,其实这也是很正常的,毕竟刚刚步入研究生科研生活。所以不要担心,这都是正常情况。下面让小编来带你来学习一下怎么获取载体序列吧!获取载体序列主要介绍NCBI/addgene/SnapGene三种序列信息检索网站。
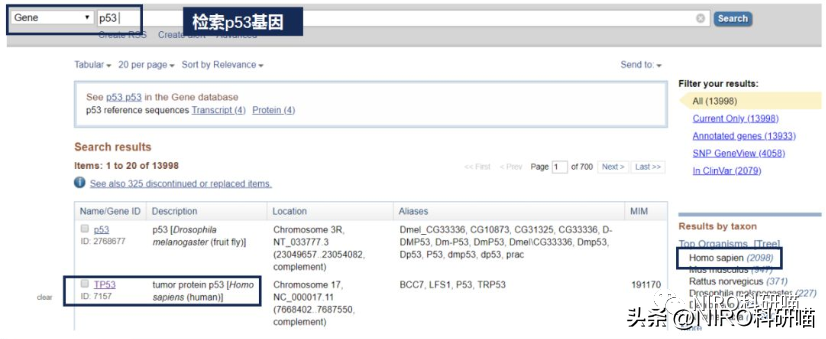
现在互联网查找基因信息是很方便的,在很多网站都可以查找到基因信息序列。而比较常用的经典基因数据库有NCBI、UCSC。下面我们就以pEGFP-C1构建humanp53CDS过表达载体为例,分享一下如何快速获取需要的序列信息。我们需要获取的序列信息有两类,一类是p53CDS也就是插入片段的序列,另一部分是载体的序列。
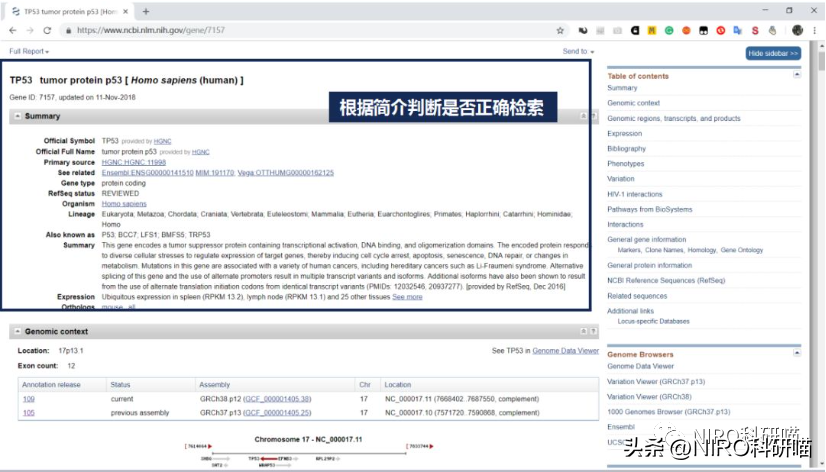
4.4.1从NCBI获取P53的CDS序列
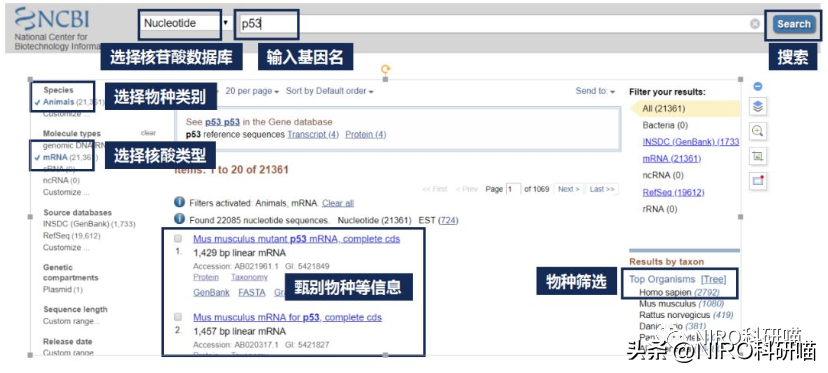
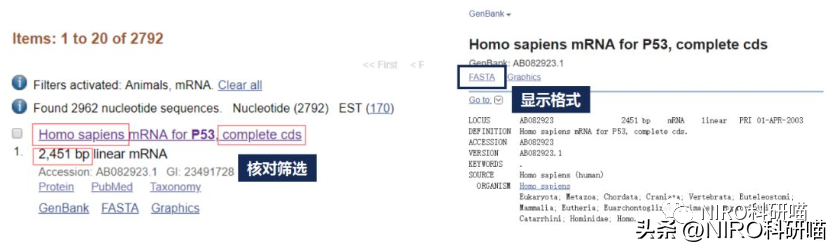
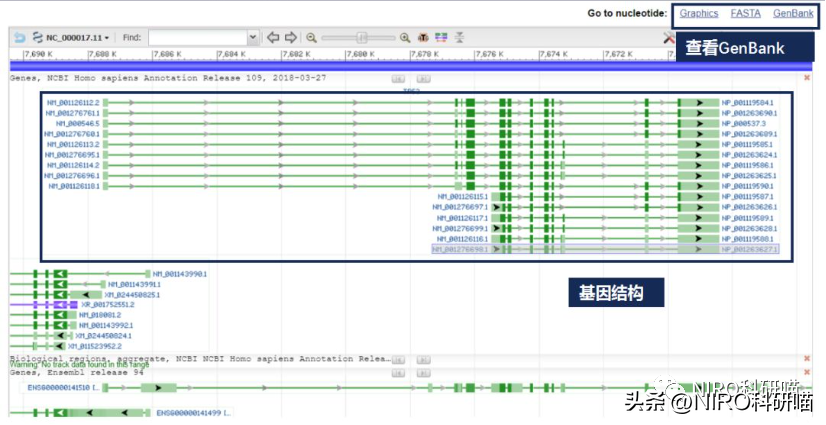
打开GenBank格式的p53序列,我们可以看到p53mRNA长度是从1bp-2451bp,其中CDS是64bp-1245bp,同样我们可以看到CDS翻译的蛋白质序列以及对应蛋白质序列的ID,方便我们进行检索,检索完成后,通过右侧“sto”进行下载。


4.4.2从Addgene获取载体pEGFP-C1
如何检索载体pEGFP-C1?通常有两种方式:
第一,通过百度搜索名称进行检索,得到的结果大概率会导向Addgene。
我们以Addgene直接检索,更快捷的得到pEGFP-C1的准确信息。打开官网,在右侧搜索栏按载体名称进行检索。为了缩小范围,我们可以在检索中进行设置,在右侧可以看到检索结果的简介。
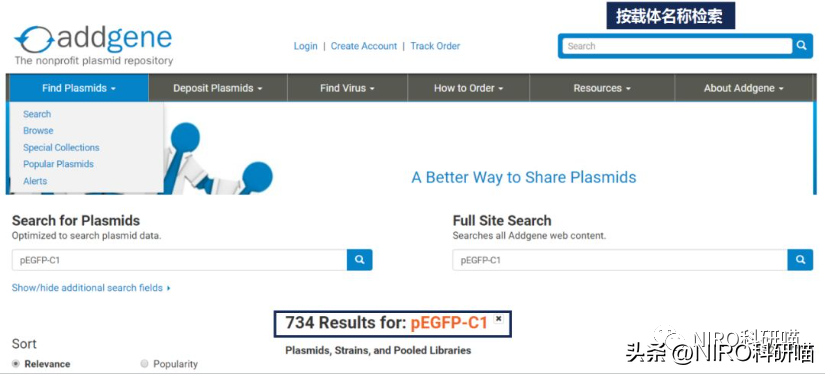
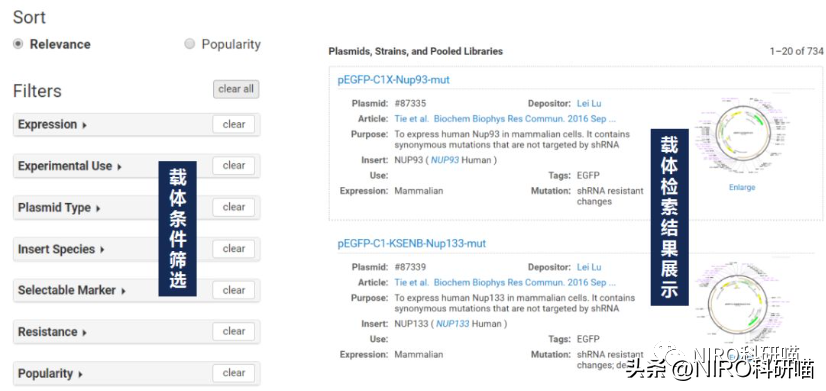
第二,通过SnapGene来对pEGFP-C1进行检索。
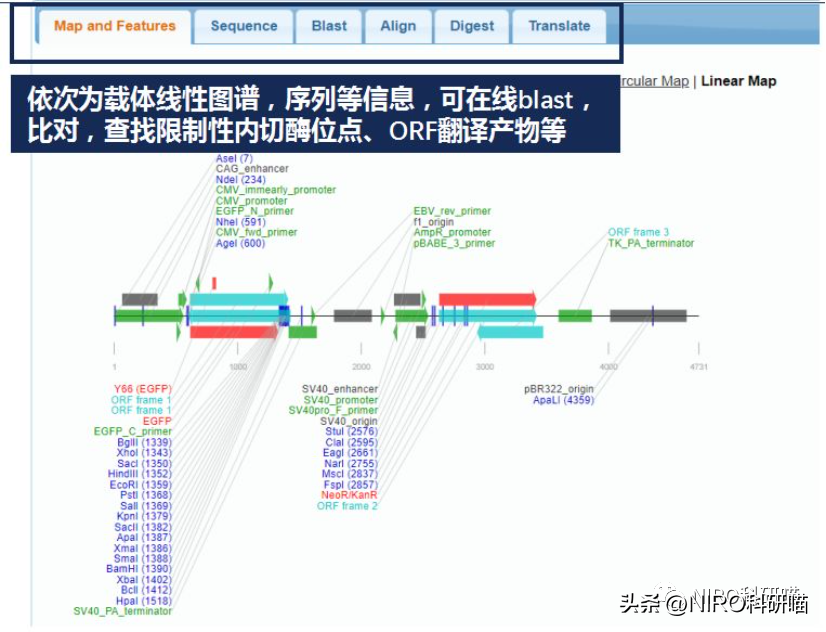
进入SnapGene官网,在主菜单栏第五个,选择质粒文件数据库,然后按照质粒的名称进行筛选,或者按照载体用途、性质来进行检索。右侧方框展示的为载体序列,查看等功能,左侧方框为pEGFP-C1载体分类。

4.4.3其他
1.Google及Google学术
检索式:质粒名+map;
质粒名+sequence+filetype:txtorfiletype:pdf
2.欧洲分子生物实验室MBL:
3.如何查找经过改造过的质粒的质粒图
4.5质粒图谱
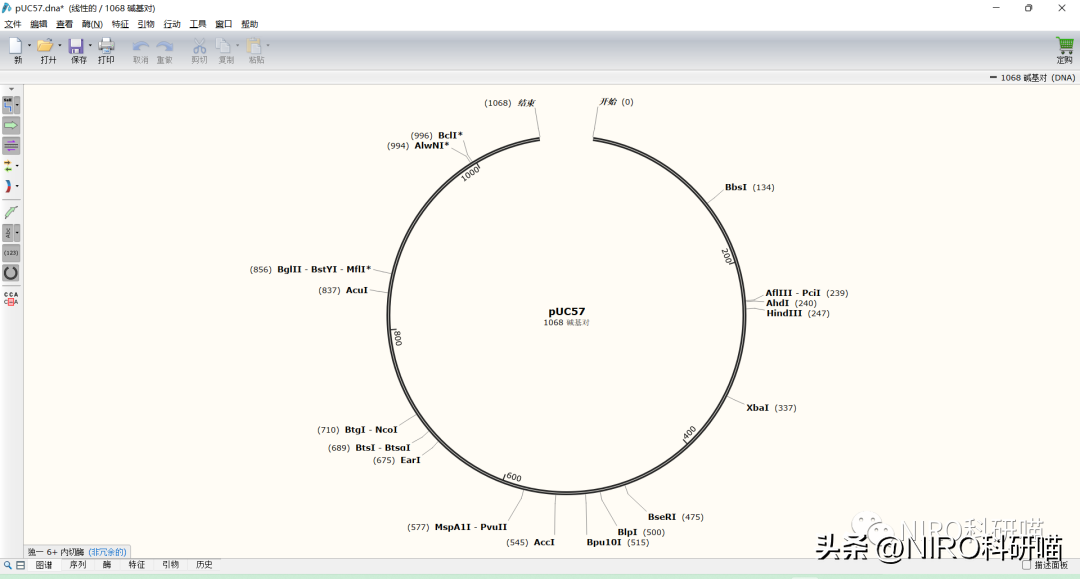
以pUC57质粒为例,怎样画出其质粒图谱呢?
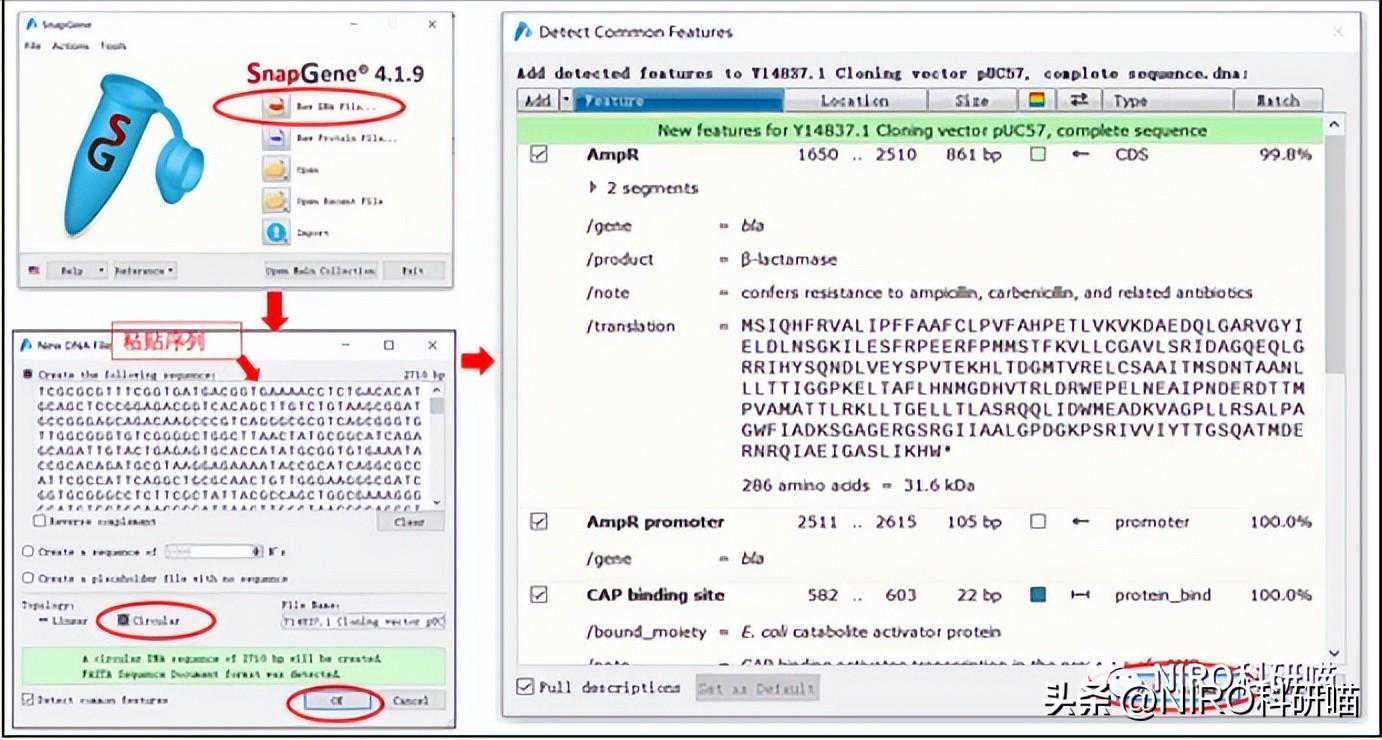
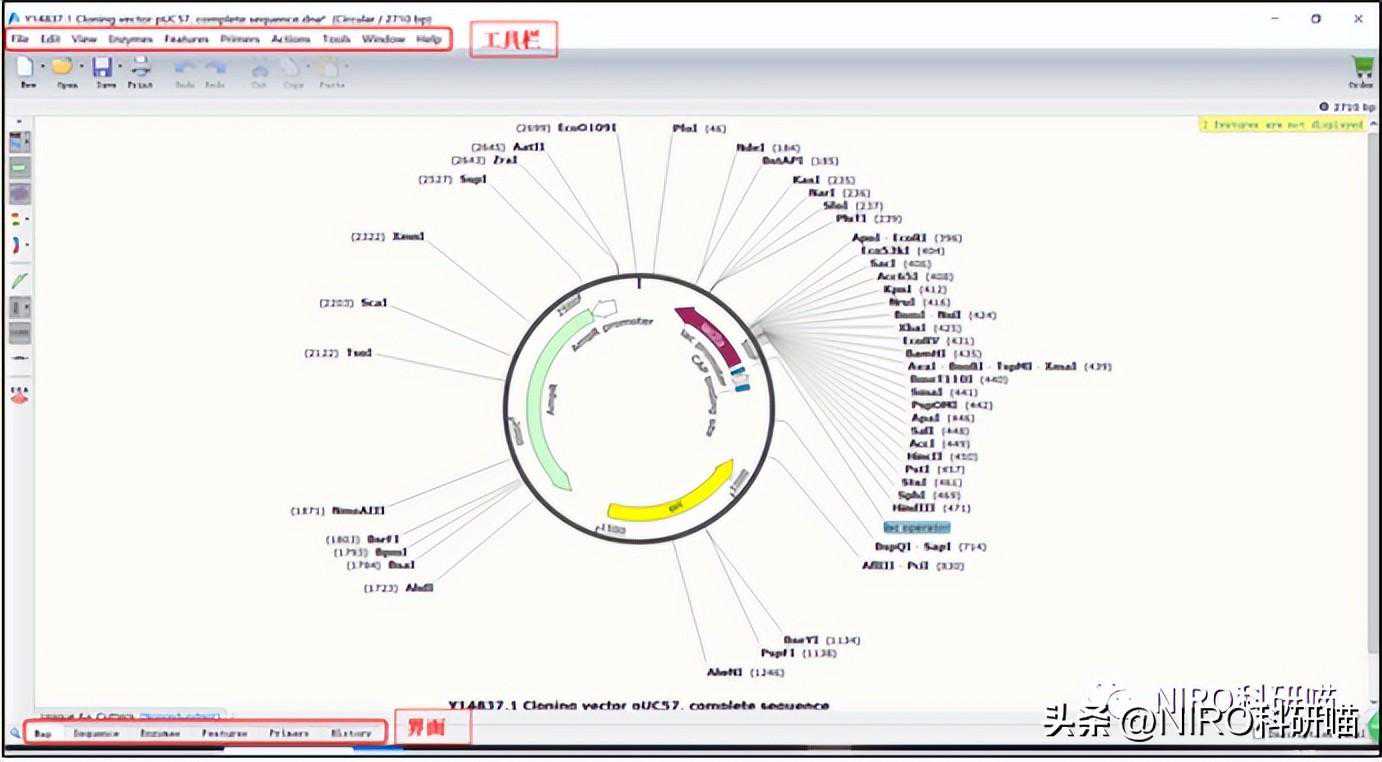
4.5质粒图谱的具体编辑方法
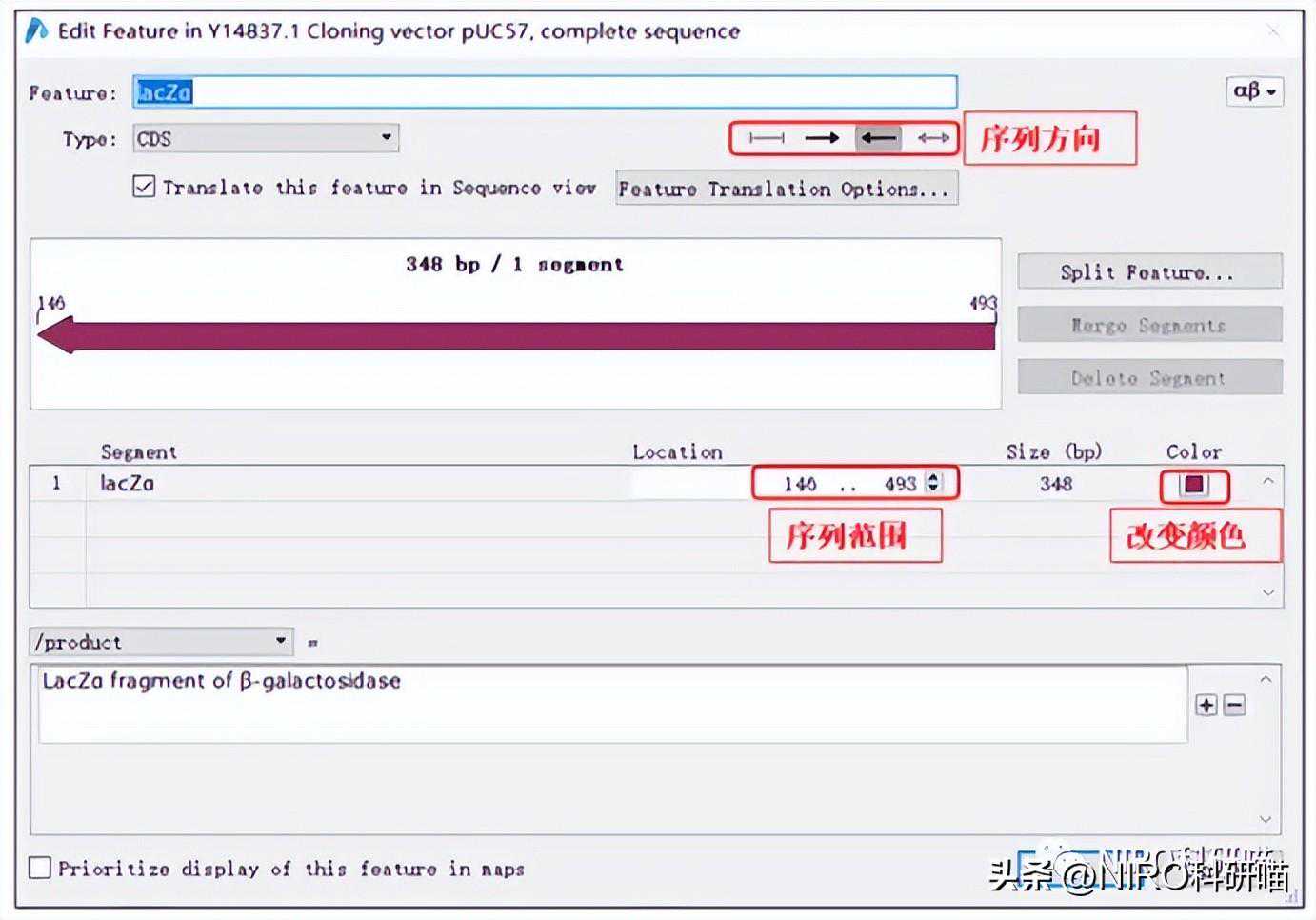
4.5.1序列标注
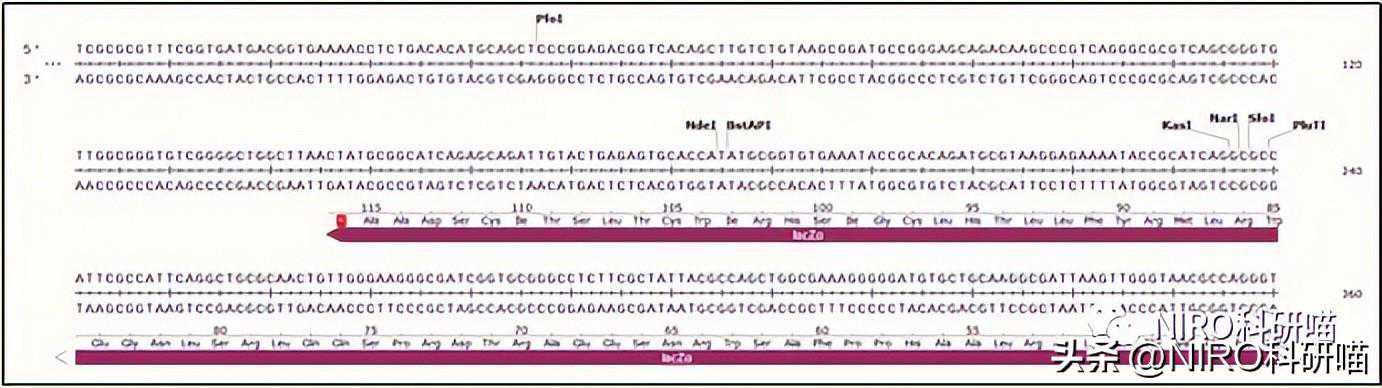
当然我们也可以自己编辑,双击lacZa的文字即可进入feature编辑界面,可以编辑序列名称、方向、范围以及颜色等信息,编辑完成后点OK,Sequence界面就会出现相应的序列注释信息。
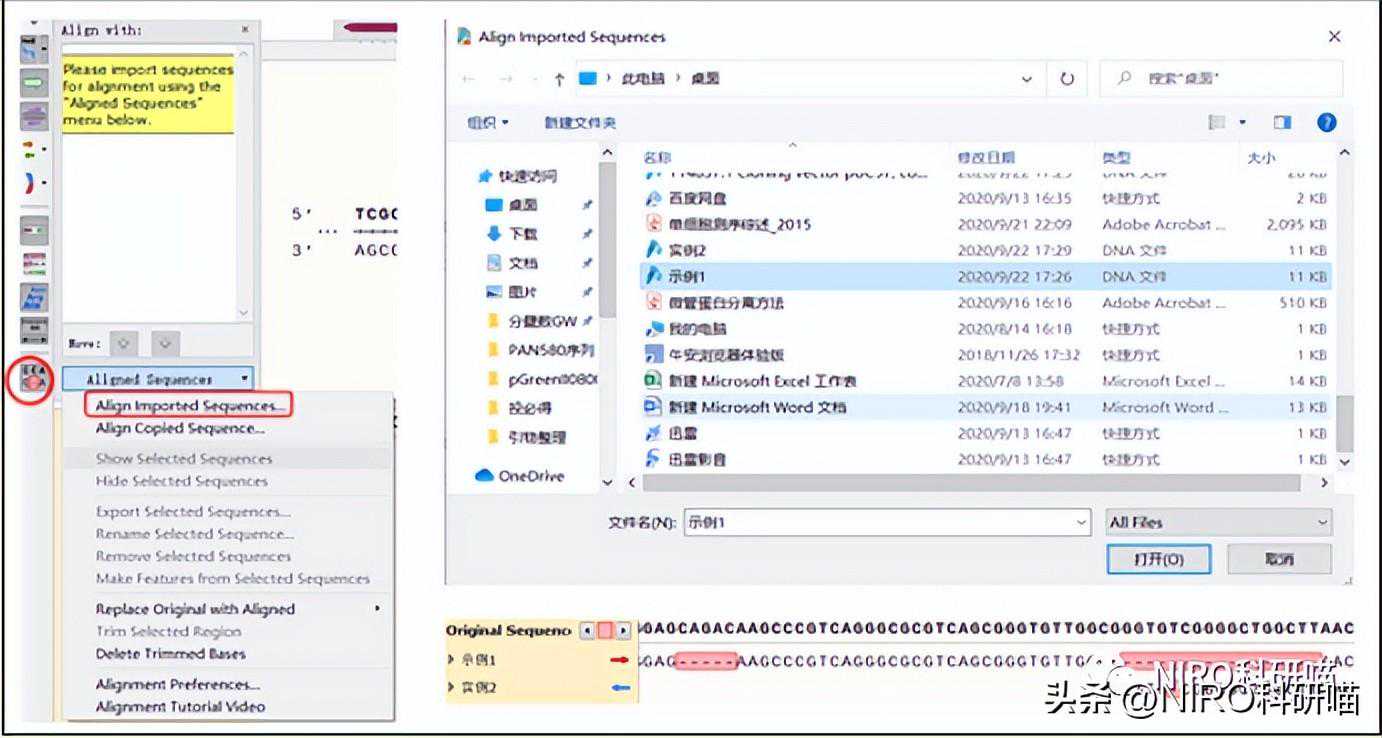
4.5.2序列比对

4.6引物设计
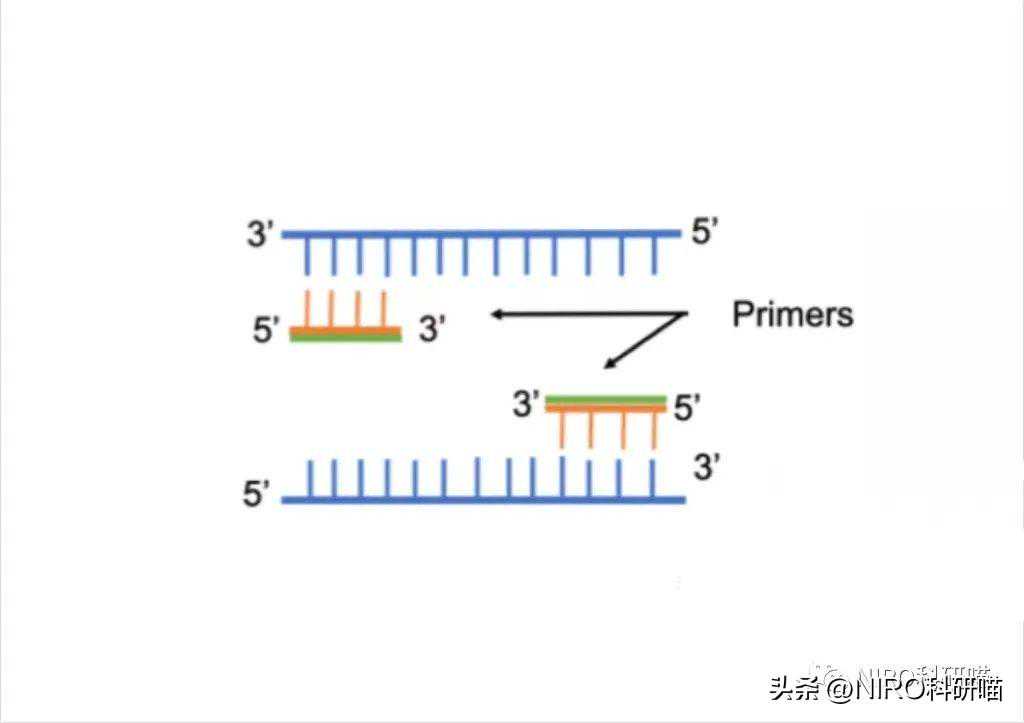
4.6.1什么是引物?
引物是一种特殊的核酸序列,用于在实验室中特异性地识别和扩增目标DNA或RNA序列。
4.6.2引物的特性
它们必须与模板分子上的序列相对应(必须与模板链互补)。
(1)引物有长短之分。短引物主要用于扩增小而简单的DNA片段,而长引物用于扩增真核基因组DNA样本。引物不宜过长(30-merprimers)或过短。过短的引物会产生不准确的非特异性DNA扩增产物,过长的引物会导致杂交速率降低。平均而言,需要扩增的DNA片段大小应该在1-10kB以内。
(2)引物结构要相对简单,不含内部二级结构,避免内部折叠。同时,还需要避免“Primer-Primer”退火,这种退火会产生引物二聚体并破坏扩增过程。设计时,如果不确定要在引物的某个位置放置什么核苷酸,可以在该位置包括多个核苷酸,称为混合位点。也可以使用基于核苷酸的分子插入物(肌苷)来代替常规核苷酸,以实现更广泛的配对能力。
4.6.3引物设计要求
个碱基的长度;
/C含量在40-60%;
c.起止于1-2G/C对;
d.熔化温度(Tm)为50-60°C;
e.引物对之间的Tm应在5°C之内;
f.引物对不应该有互补区域;
4.6.4SnapGene引物设计操作
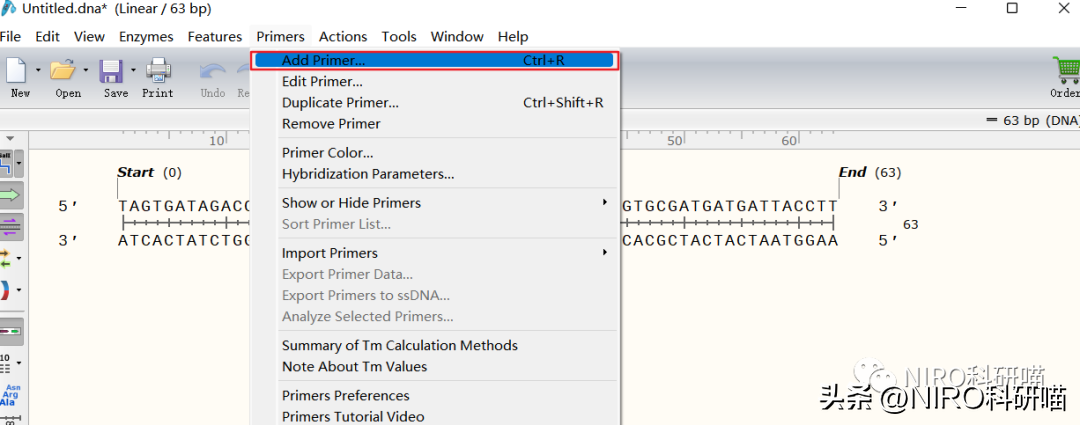
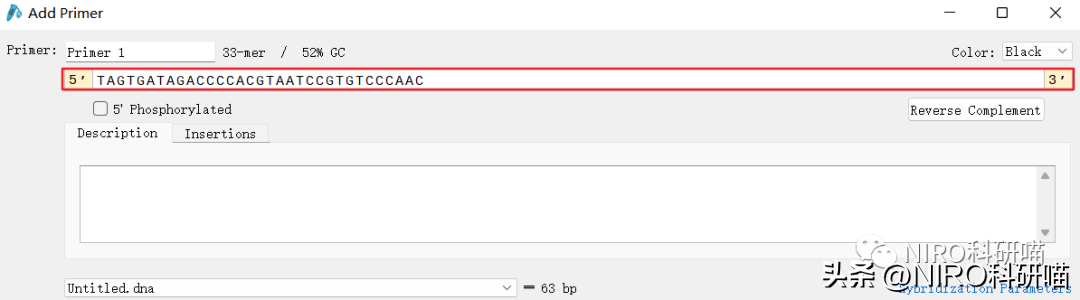
4.6.4.2选择绑定位点
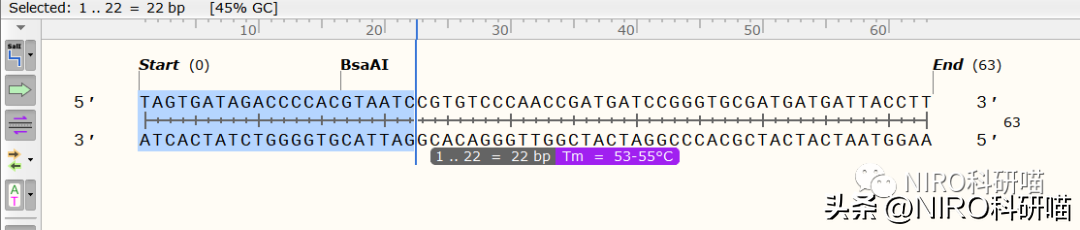
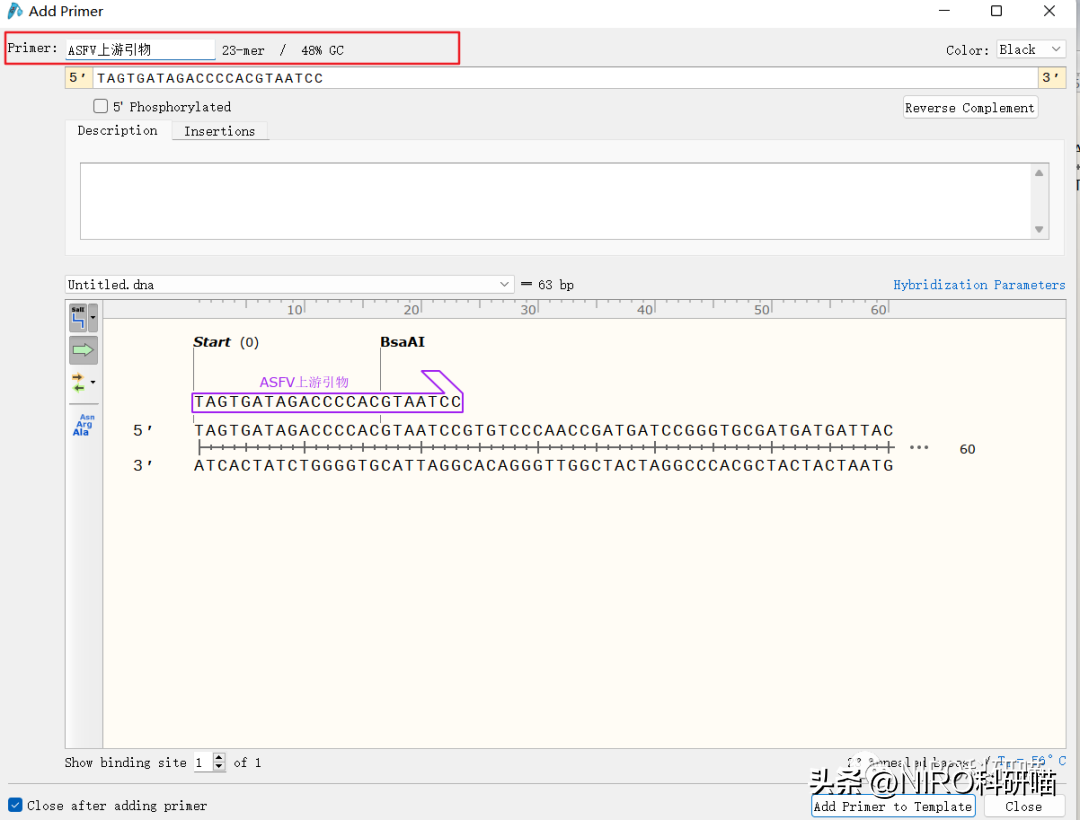
4.6.4.3引物命名
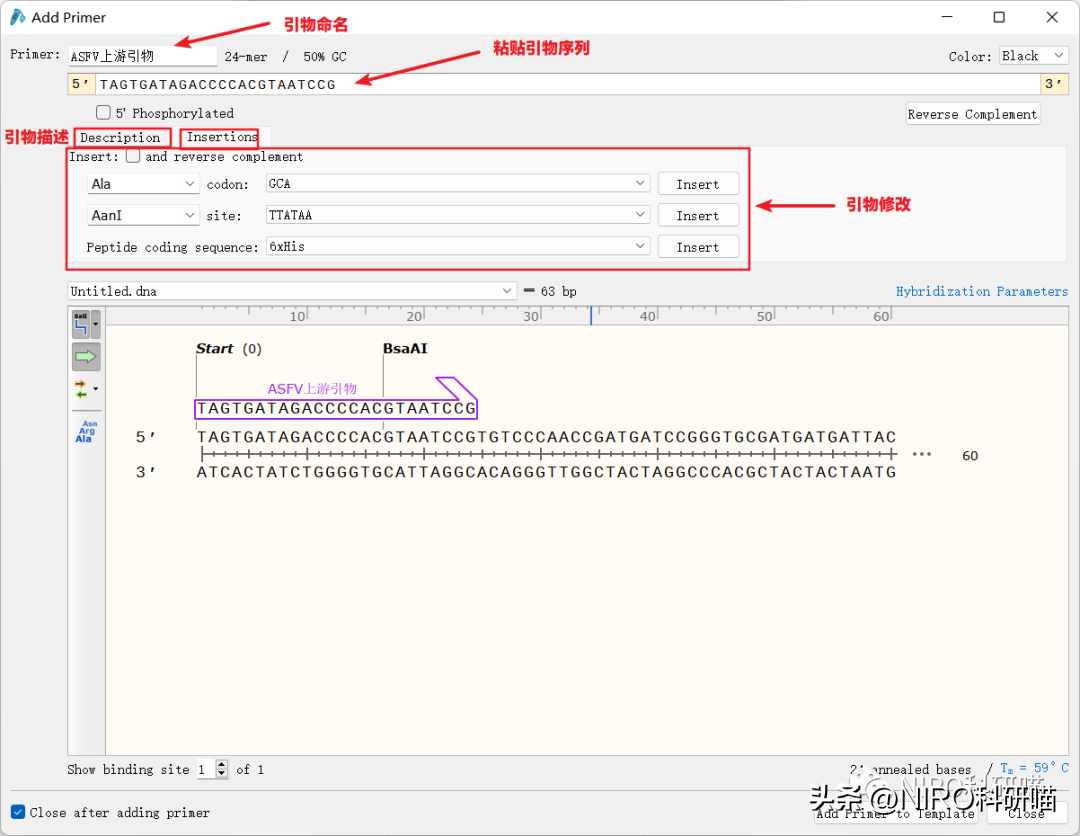
4.6.4.4修改引物
4.6.4.5查看结合部位和溶解温度
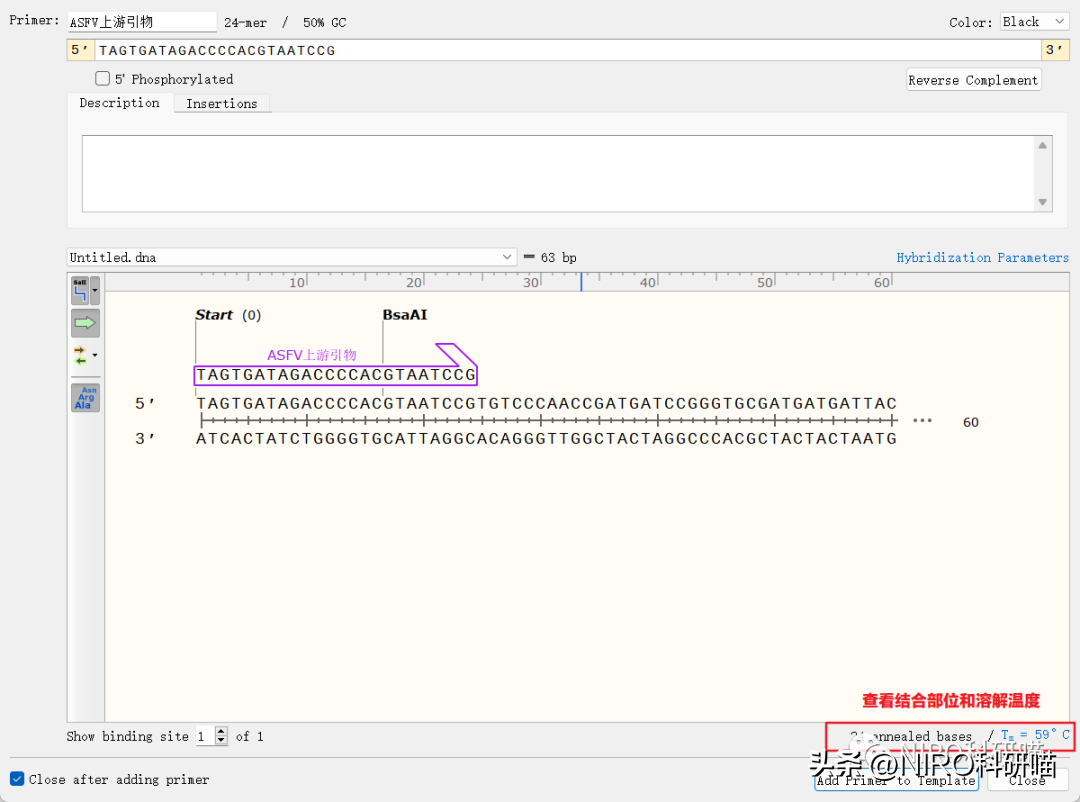
4.6.4.5查看设计完的引物
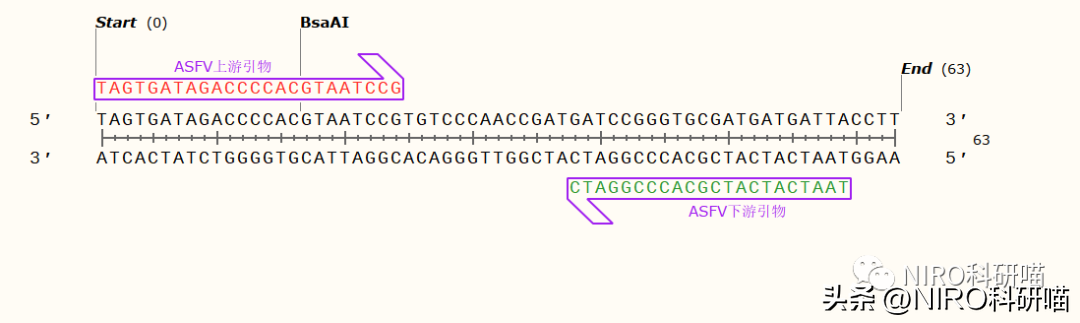
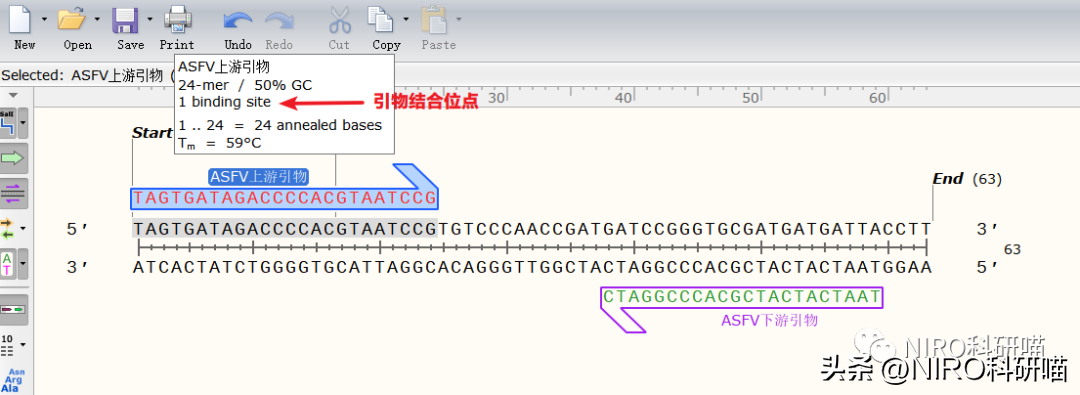
4.6.4.6查看引物的结合位点
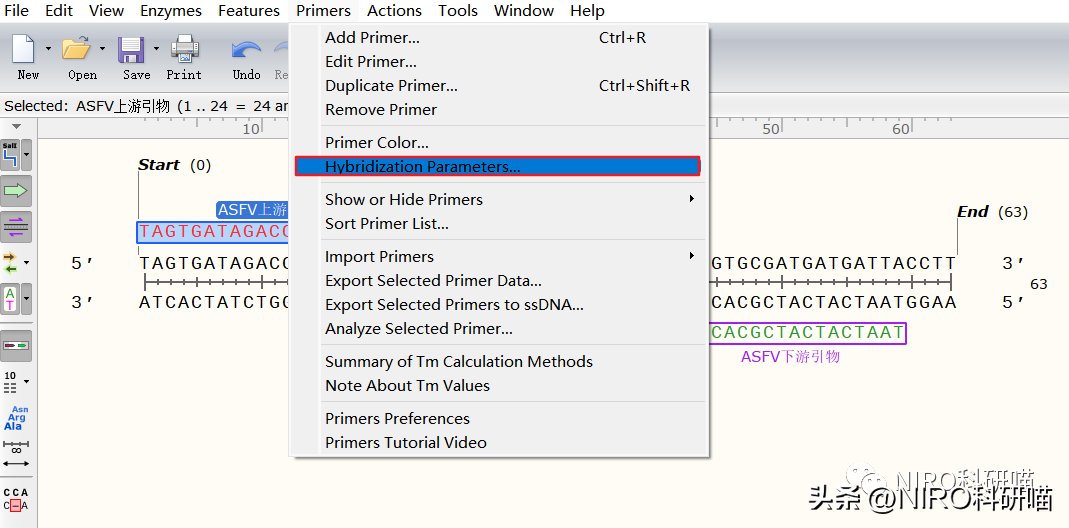
4.6.4.7调整杂交参数
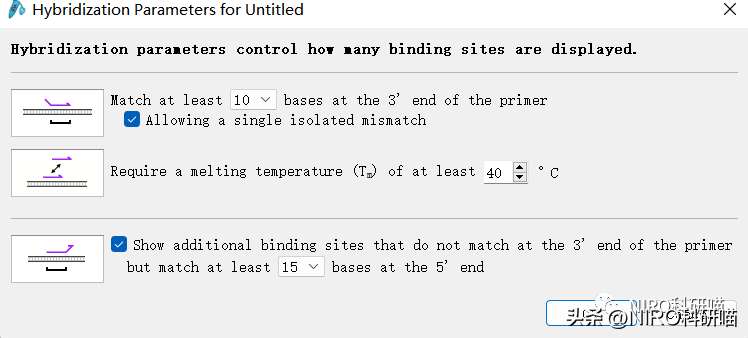
4.6.4.8隐藏/显示引物
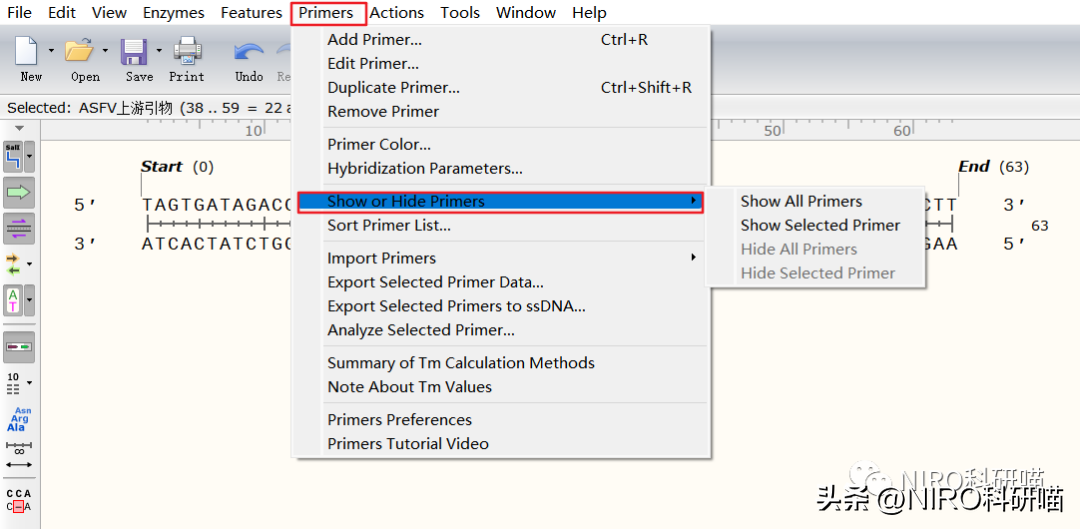
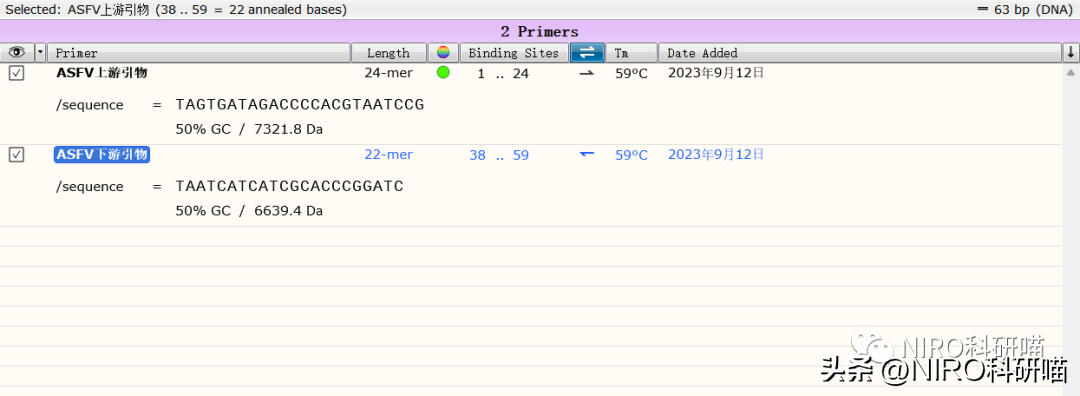
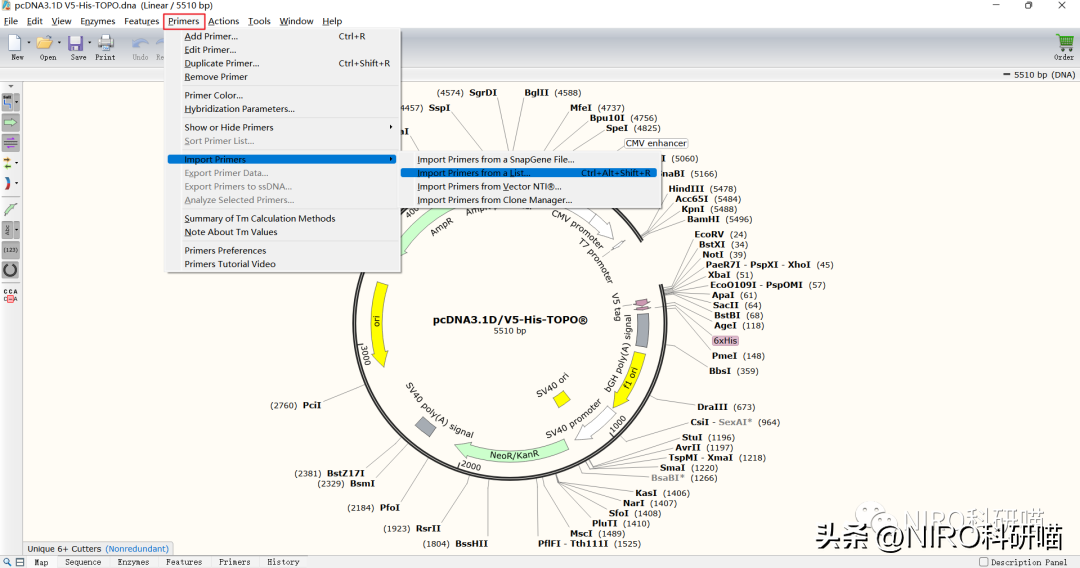
4.6.4.9导入引物,如何将引物从一个文件夹导入另一个文件夹
1.打开目标文件夹
打开将接收导入的引物的文件。SnapGene将仅导入可与目标文件序列杂交的引物。如果目标文件已包含引物,则SnapGene默认情况下不会复制现有的引物,但会提供导入重复的引物的选项。
一个*.dna源文件
一个*.txt*.csv,*.tsv或*.rtf格式的列表
一个VectorNTI®数据库或oligo文件
一个CloneManager引物文件
下面分别讲解一下如何用SnapGene操作以上四种导入引物的步骤。
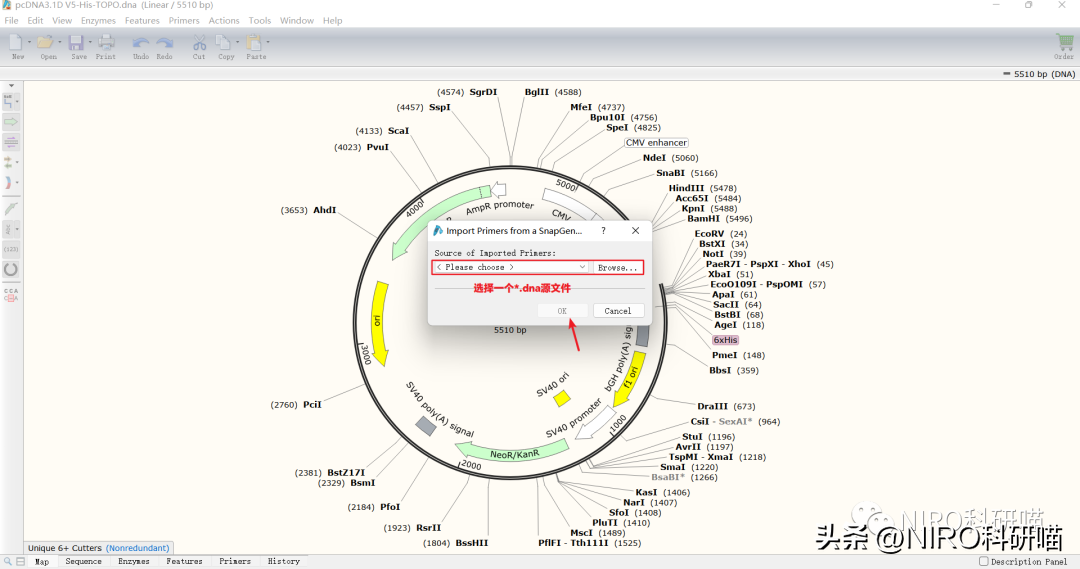
(1)导入一个*.dna源文件

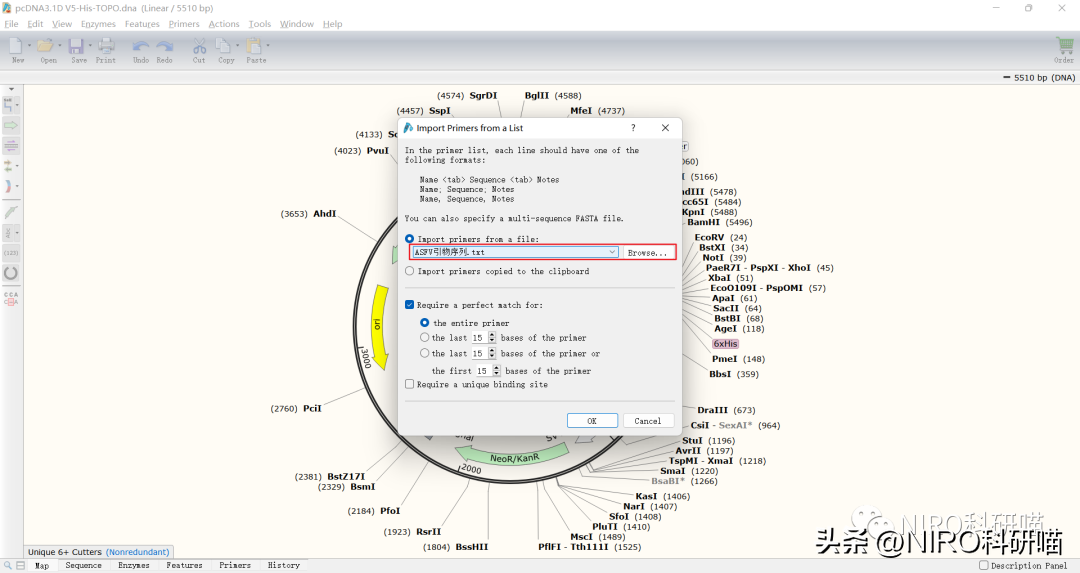
(2)导入一个*.txt*.csv,*.tsv或*.rtf格式的列表
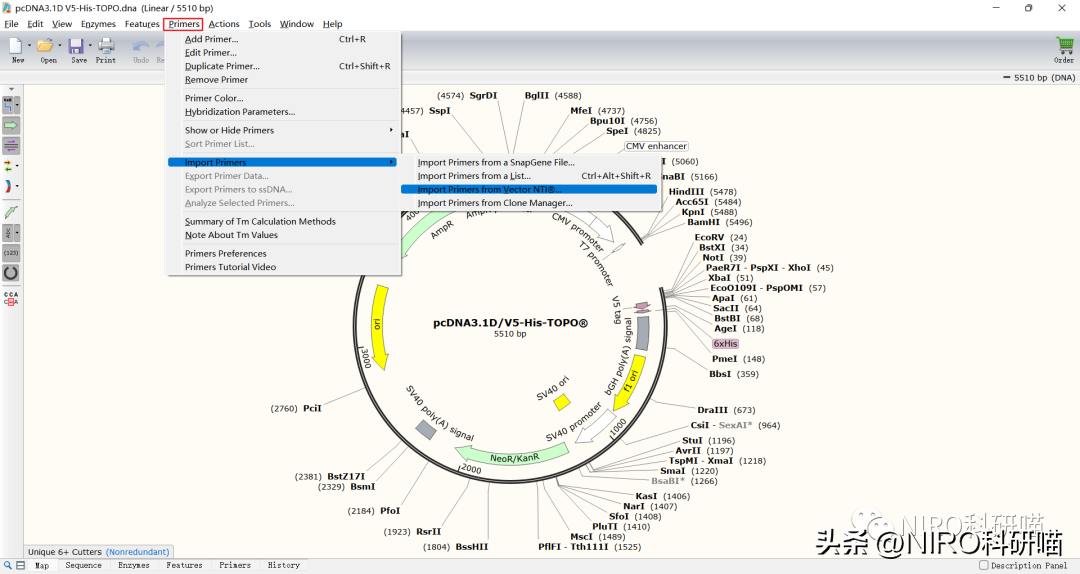
(3)导入一个VectorNTI®数据库或oligo文件
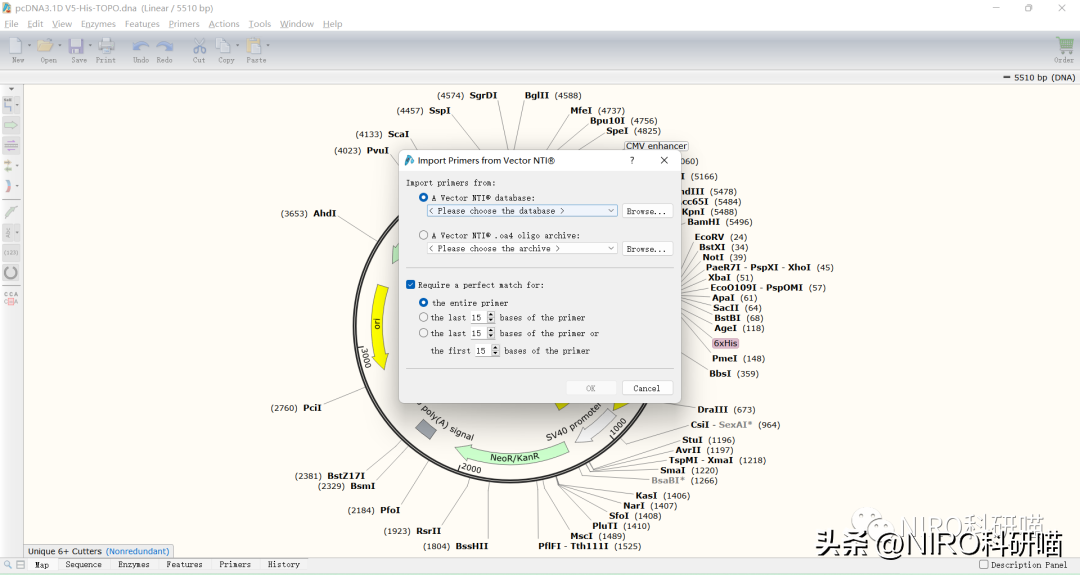
(4)导入一个CloneManager引物文件
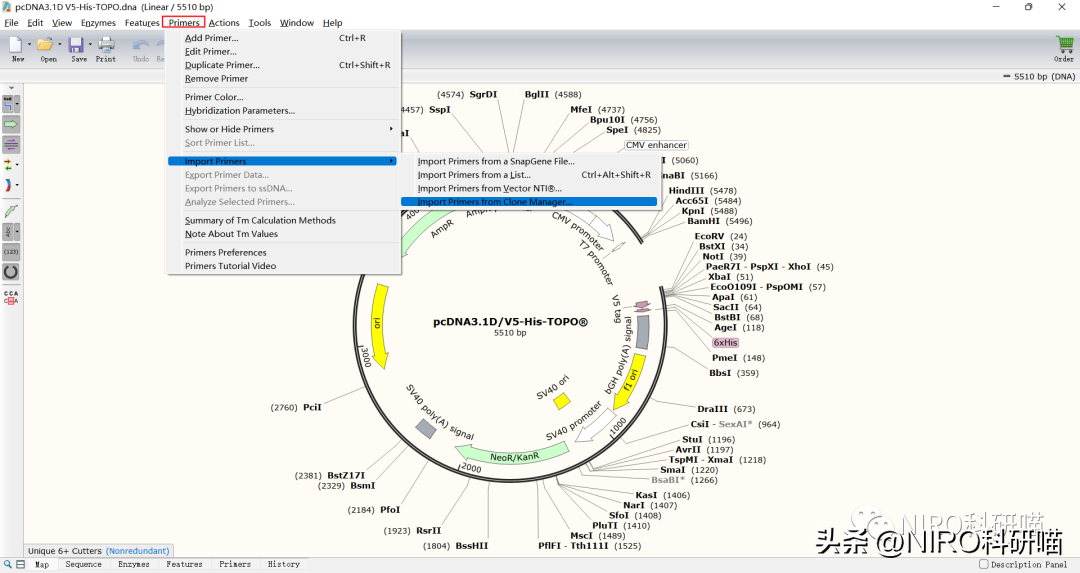
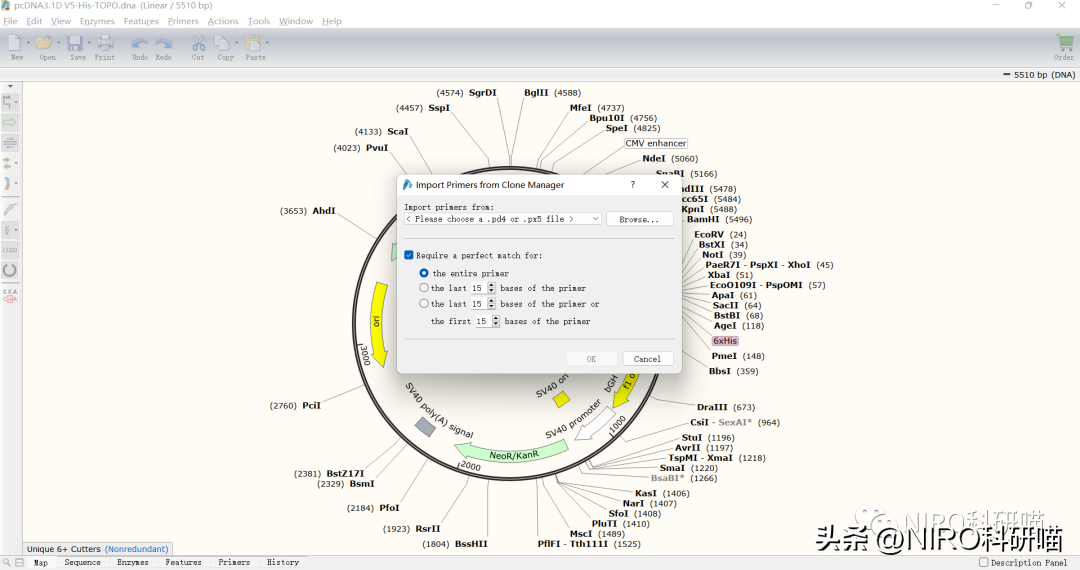
2.查看AvailablePrimersList
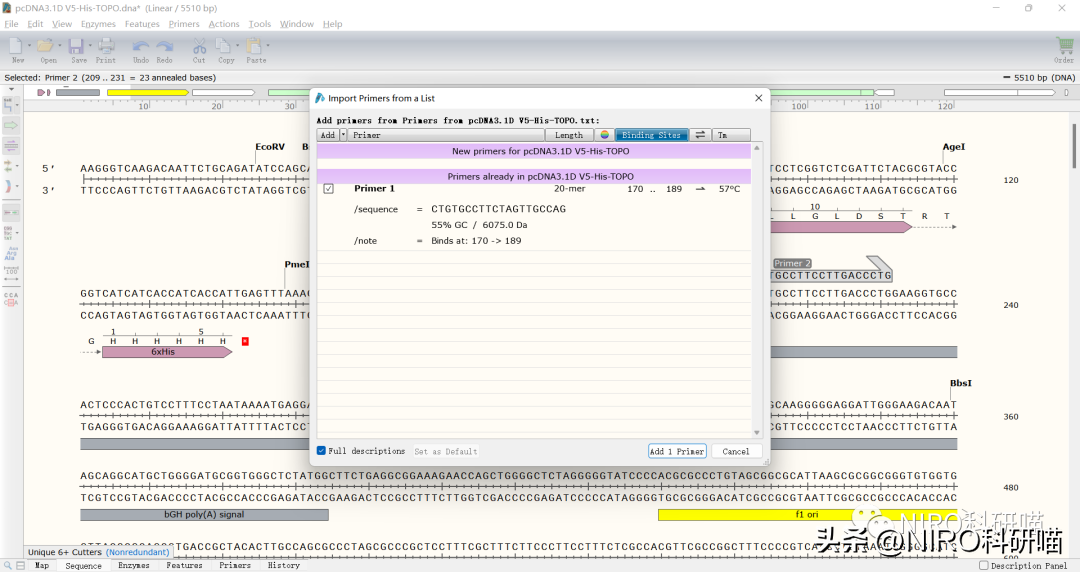
3.指定添加哪些引物
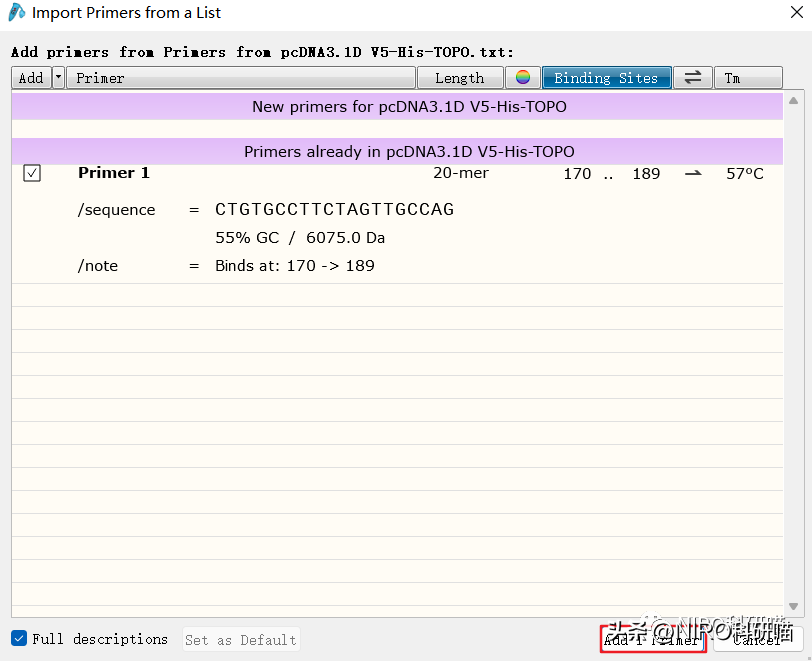
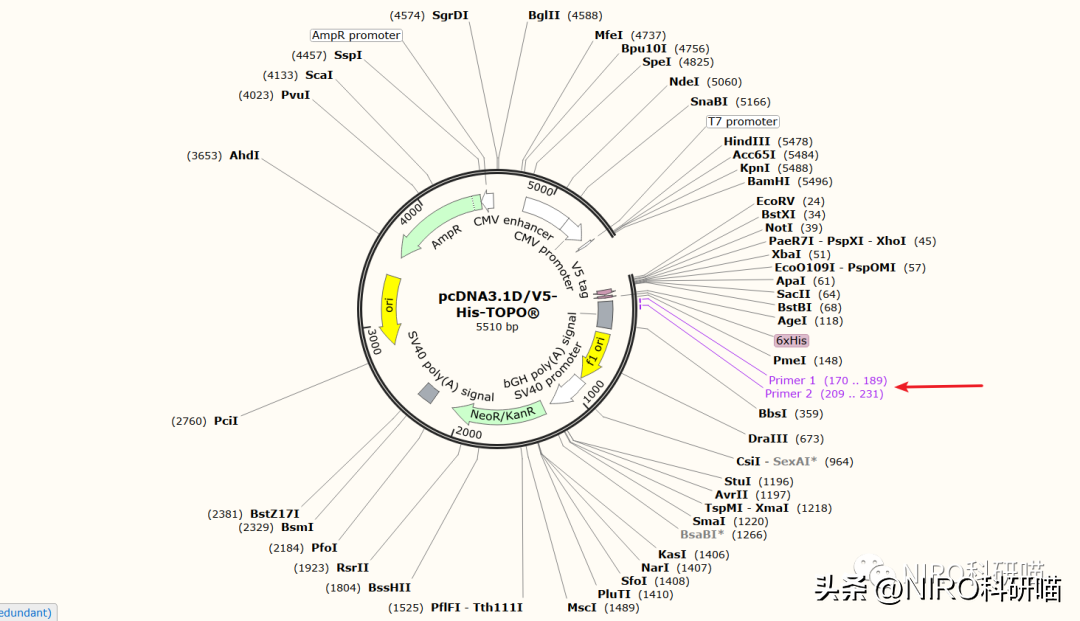
4.查看导入的引物
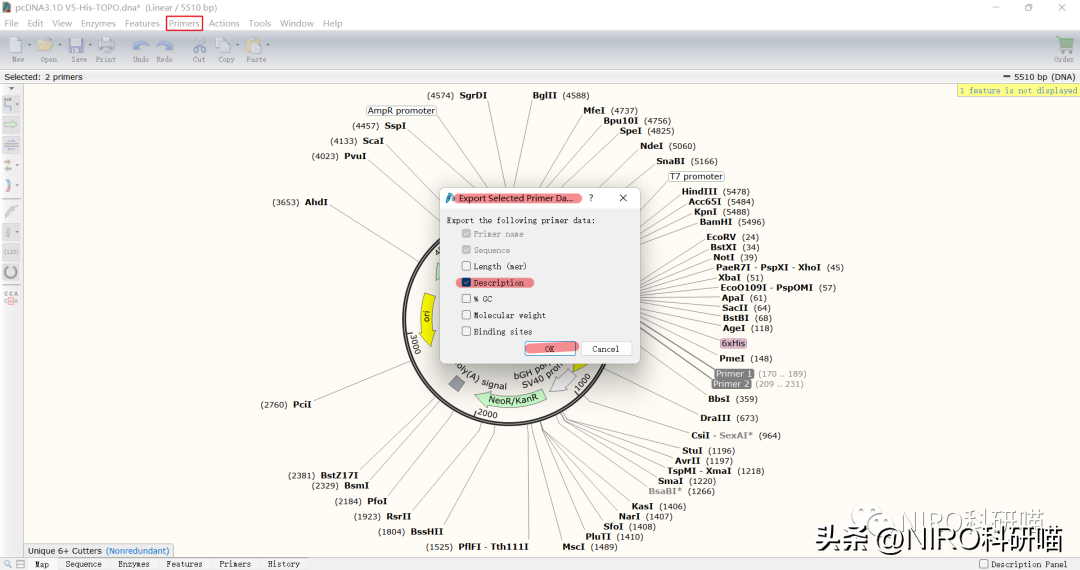
5.导出引物序列
4.7Actions里面常用的功能介绍
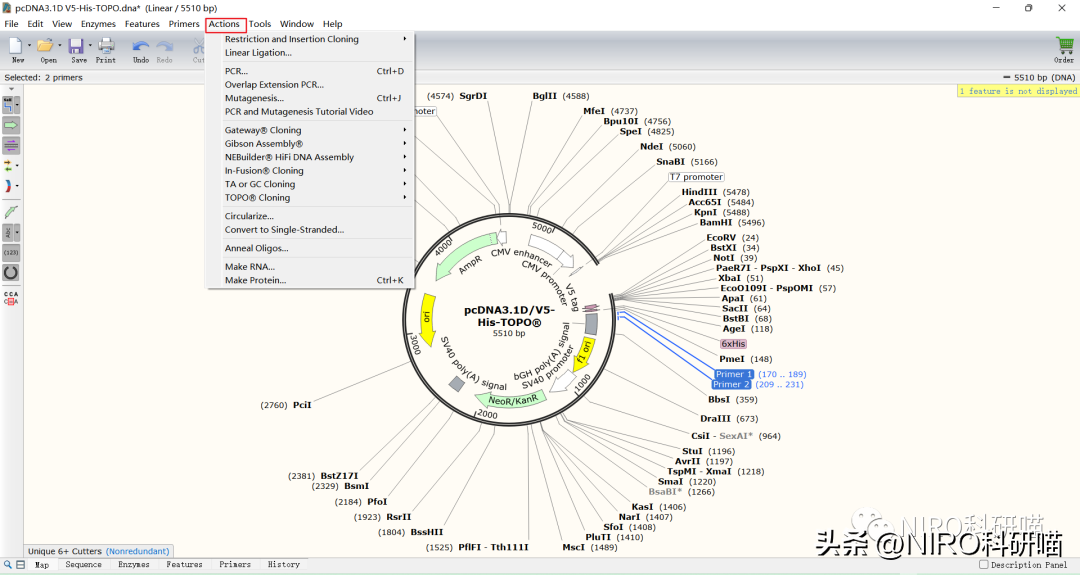
在介绍这里常用的功能,小编换SnapGene中文版进行讲解,为什么呢?主要考虑到很多小伙伴对这个软件还不熟悉,如果用SnapGene英文版本,会到增加一定的难度,因此小编在这里就选择SnapGene中文版进行讲解,确保大家可以一次性学会!!!
下面先介绍“Actions”在SnapGene中文版对应的名称“行动”。那么,在“行动”中有哪些功能呢?我们看下图即可。
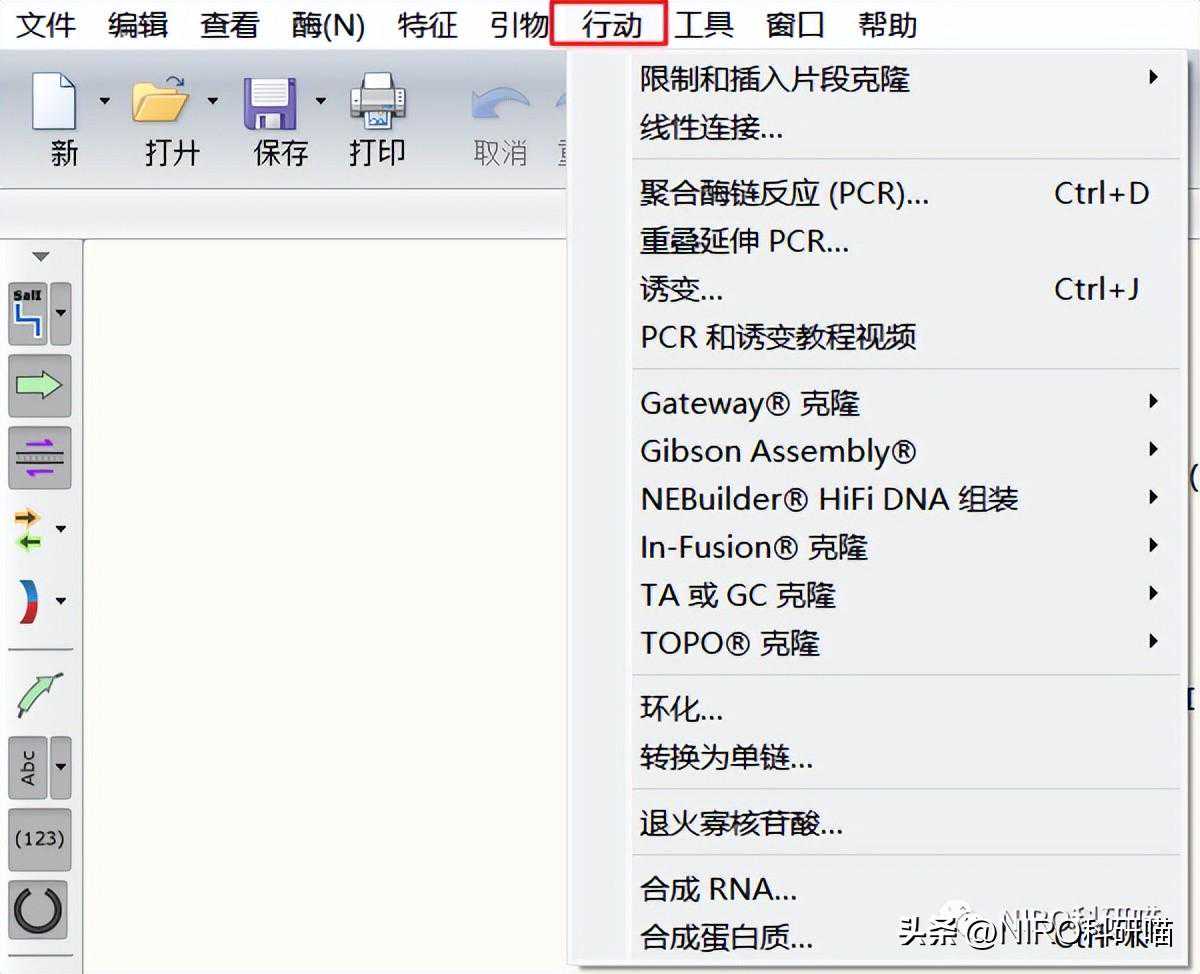
4.7.1限制和插入片段克隆
这个功能在SnapGene软件第三期内容已经介绍过了。所以这期就不进行讲解了!!!有不知道的小伙伴,可以到SnapGene软件第三期去学习。
4.7.2线性连接、环化、退火寡核苷酸以及氨基酸序列
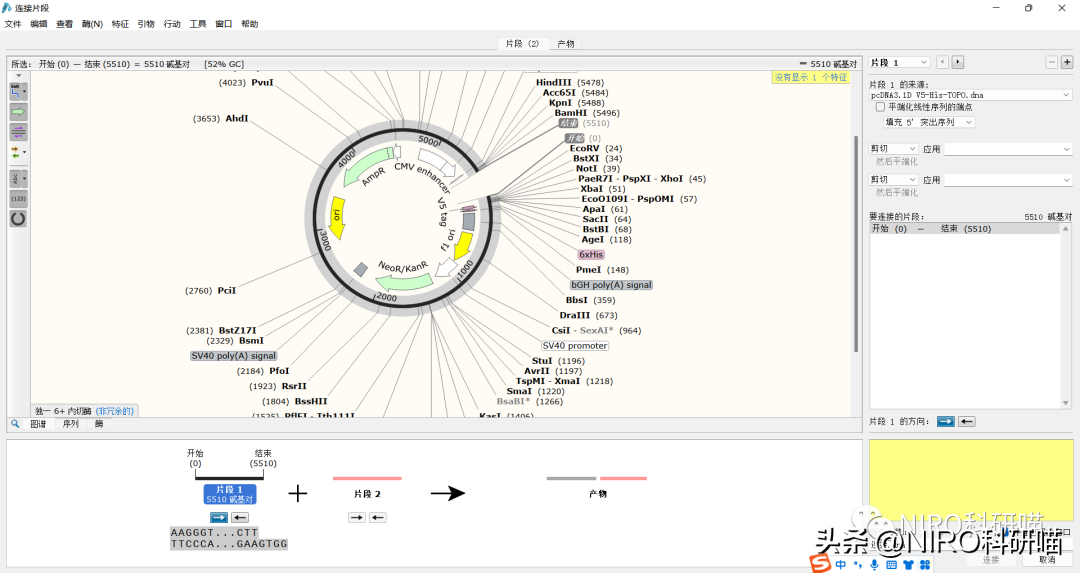
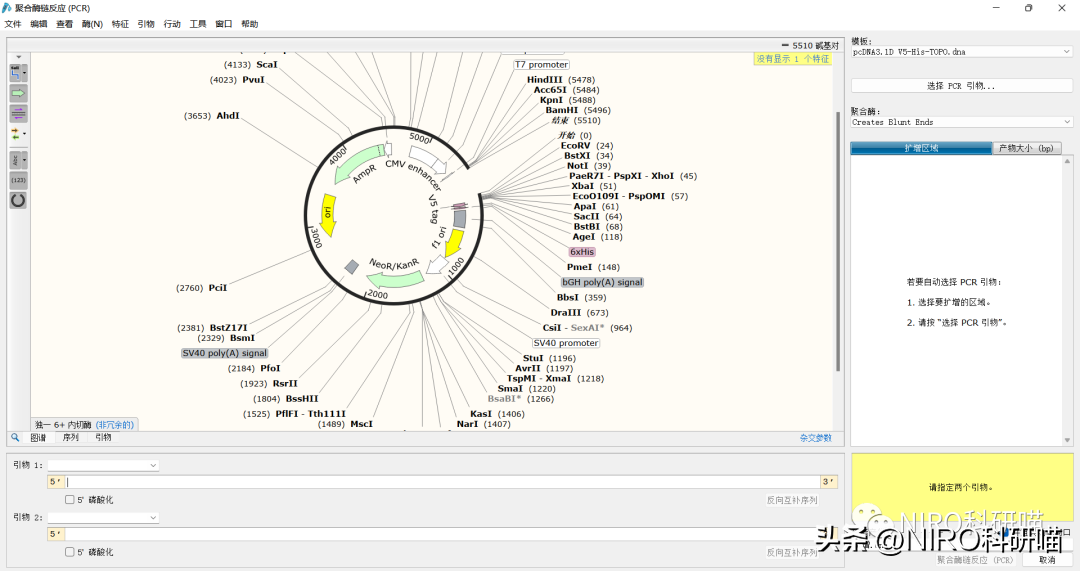
4.7.3聚合酶链反应(PCR)
聚合酶链式反应(PCR)是一种用于放大扩增特定的DNA片段的分子生物学技术,它可看作是生物体外的特殊DNA复制,PCR的最大特点是能将微量的DNA大幅增加。
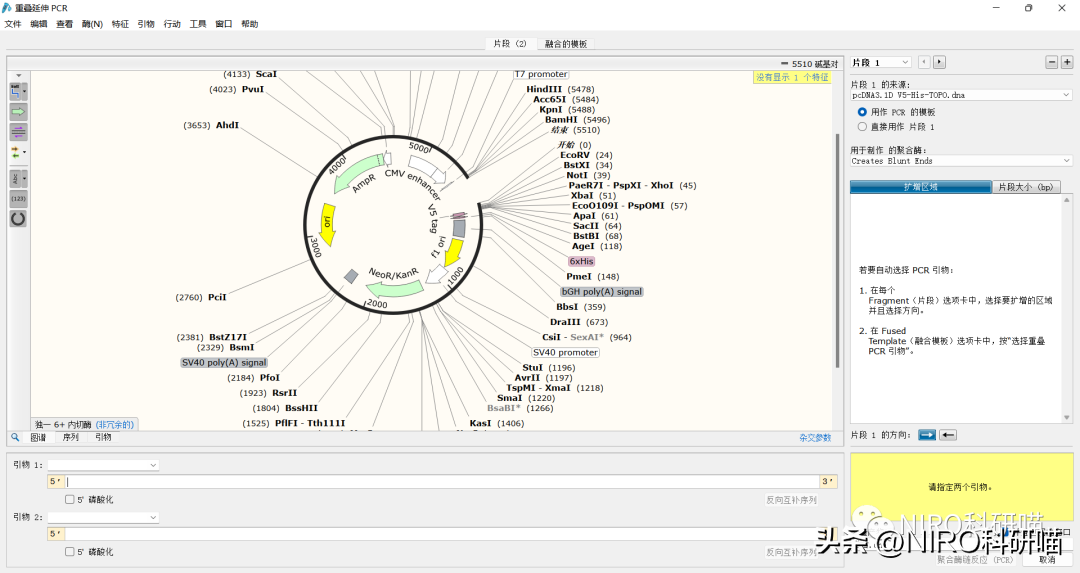
4.7.4重叠延伸PCR
重叠延伸PCR(OverlapExtensionPCR),又称为SOE-PCR(SplicingbyOverlapExtensionPCR),是一种用于构建DNA重组或基因突变的分子生物学技术。它通过两轮PCR反应,将两个或多个DNA片段的重叠部分连接起来。
4.7.5Gateway克隆
Gateway克隆是一种高效的DNA重组技术,用于构建重组DNA。它基于Gateway技术,利用特殊的重组酶将DNA片段插入到预先构建好的载体中。
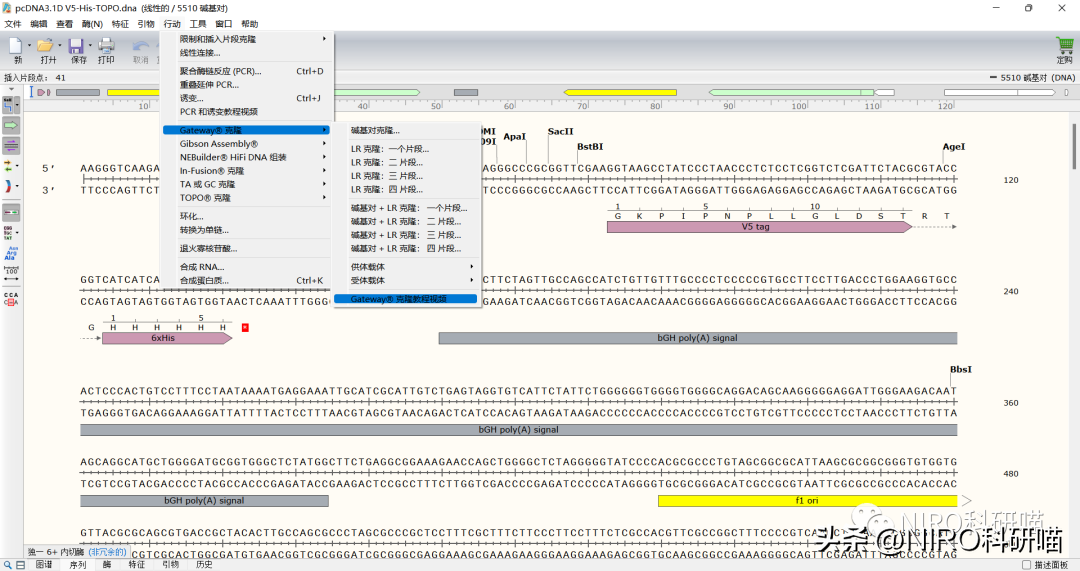
4.7.6GibsonAssembly
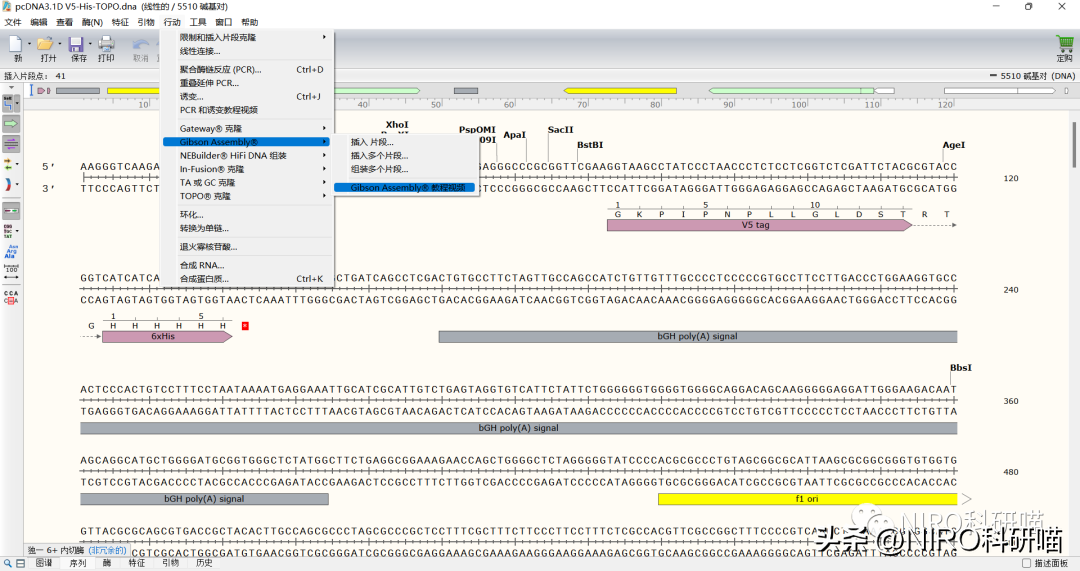
GibsonAssembly是一种高效的DNA片段连接方法,用于构建重组DNA。它基于酶体外的DNA连接酶活性,不需要限制性内切酶和连接酶,因此避免了酶切和连接步骤中可能出现的限制性酶位点缺失和连接错误的问题。
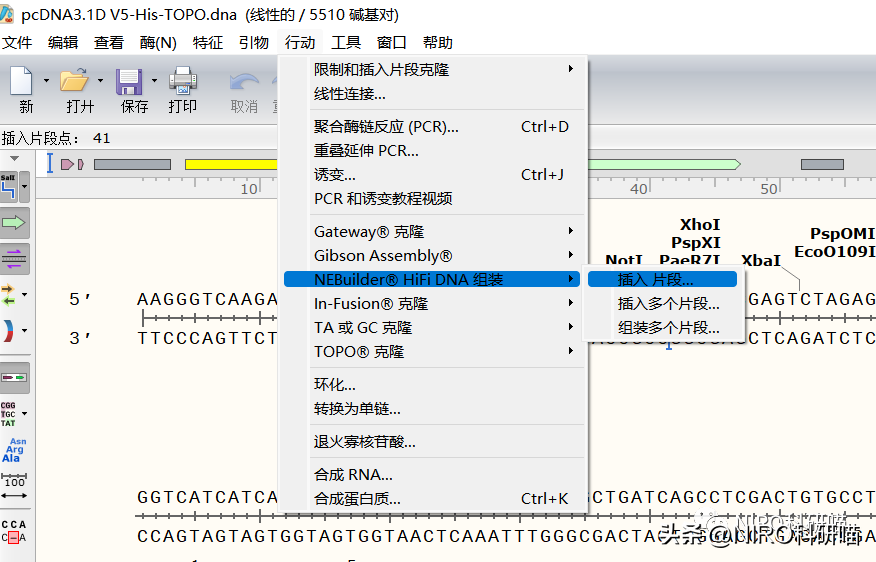
4.7.7NEBuilderHiFiDNA组装
NEBuilderHiFiDNA组装是一种高效的DNA片段连接方法,用于构建重组DNA。它基于NEBuilderHiFiDNAAssemblyMasterMix,该酶混合物包含了高效的DNA连接酶、高保真度的DNA聚合酶和缺失校正酶。
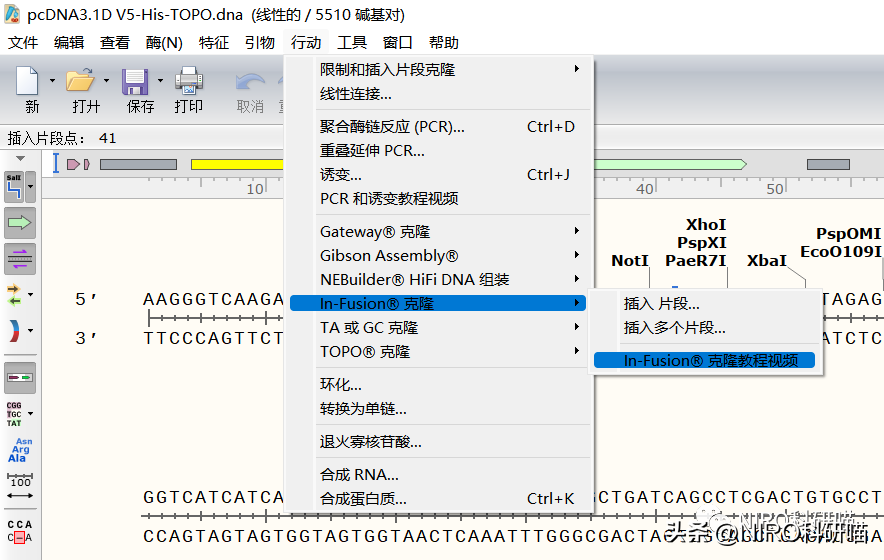
4.7.8In-Fusion克隆
In-Fusion克隆是一种高效的DNA重组技术,用于构建重组DNA。它基于酶体外的DNA连接酶活性,不需要限制性内切酶和连接酶,因此避免了酶切和连接步骤中可能出现的限制性酶位点缺失和连接错误的问题。
4.7.9TA或GC克隆
TA克隆和GC克隆都是常见的DNA克隆技术。TA克隆是一种利用特殊的TA酶切酶和TA克隆载体进行的克隆技术,它适用于克隆PCR产物或其他具有A尾的DNA片段。GC克隆则是一种利用GC富集的克隆载体和GC富集的DNA片段进行的克隆技术,它适用于克隆GC含量高的DNA片段。两种克隆技术各有优缺点,选择哪种克隆技术应根据具体的实验需求进行决定。
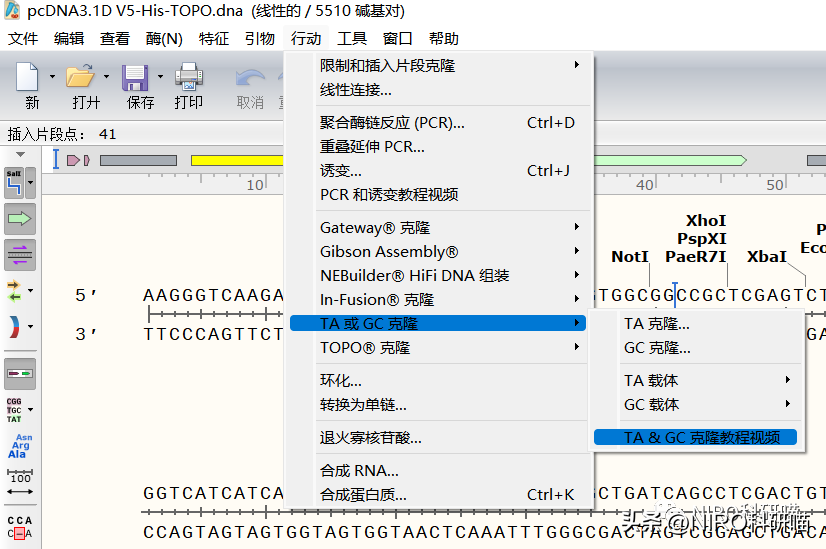
4.7.10TOPO克隆
TOPO®克隆(基于拓扑异构酶的克隆技术)不需要限制性内切酶或外源连接酶,从而提供了将新的PCR产物克隆到质粒中的极其简便快捷的方法。该技术依赖于互补的碱基对腺嘌呤(A)和胸腺嘧啶(T)的基本能力来杂交并形成氢键。
4.8模拟琼脂糖凝胶电泳
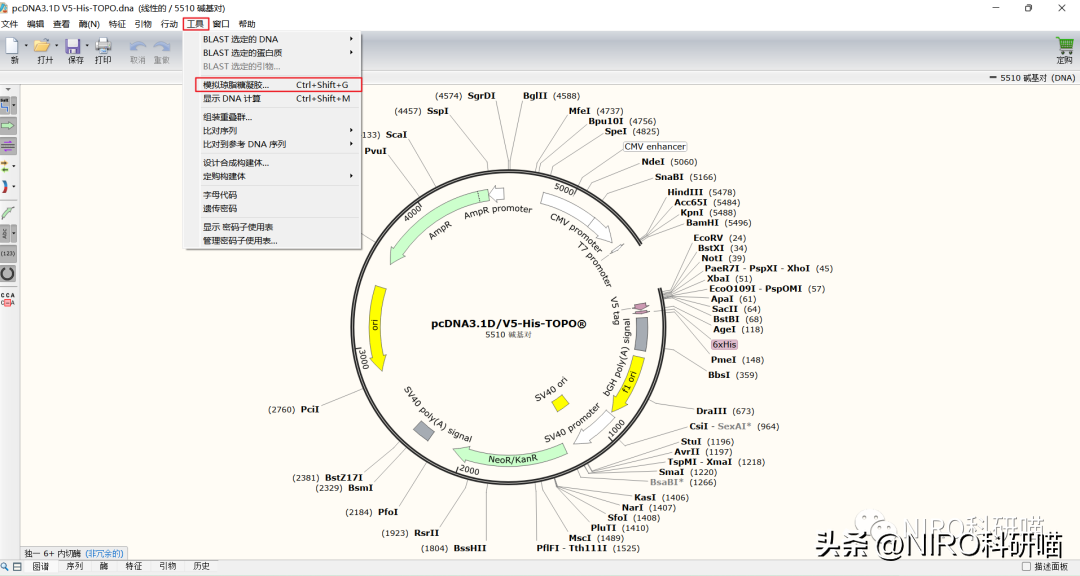
比如,这里我们对插入的目的基因进行电泳检测。
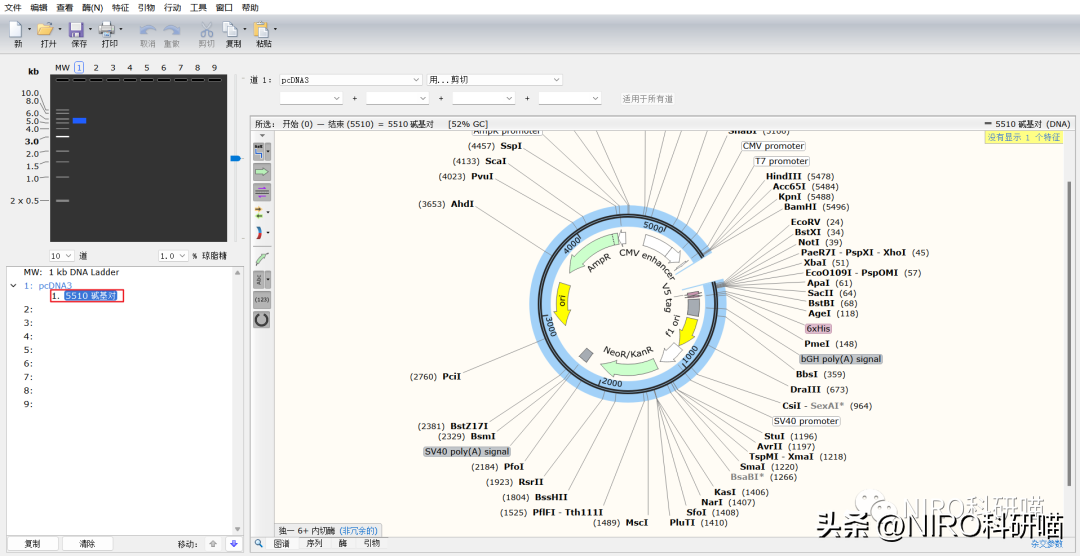
最后,保存质粒图片,或者保存为SnapGene文件,以便日后再编辑。


5
SnapGene常见问题
5.1质粒提取浓度高低不一的问题
作为一个实验小白,在很多时候提取质粒所得造成的浓度差异是很大的,有的浓度高达500-600ng/μL,有的才只有几十。小编也时常会遇到这种情况,为什么用的都是一样的东西(试剂盒和菌株),但质粒提取的浓度为什么会有这么大的差异呢?
要解决这个问题,我们首先要了解质粒!!!
1.拷贝数
对于质粒载体,拷贝数是我们最关心的特性之一。实际上,每个细菌中的质粒的拷贝数主要决定于质粒本身的复制特性。按照复制性质,可以把质粒分为两类:
严紧型质粒:当细菌染色体复制一次时,质粒也复制一次,每个细菌内只含1~2个质粒。
松弛型质粒:当细菌染色体复制停止后仍然能继续复制,每一个细菌内一般含20个左右质粒拷贝。这些质粒的复制是在寄主的松弛控制之下的,每个细菌中含有10-200份拷贝,如果用一定的药物处理抑制寄主蛋白质的合成还可使质粒拷贝数增至几千份。
当然,恒定的拷贝数与质粒复制控制系统、质粒的大小及培养条件有关!那么到这里你可能会问,高拷贝质粒我们可以多提一点质粒,低拷贝数的质粒有什么作用呢?确实,低拷贝数的质粒用途不是十分广阔,主要用于以下两点:
(1)高拷贝数的质粒往往不稳定,进行大片段克隆或者带有毒性DNA克隆时会用低拷贝。
(2)质粒的扩增会占用大量资源,当载体用于表达或者其他用途时,也会使用上低拷贝质粒。

2.质粒的接合转移与穿梭质粒
质粒的接合转移:是细菌遗传物质转移的一个重要方式。在质粒转移过程中,供体菌和受体菌通过结合作用紧密接触,质粒从供体细胞向受体转移,同时进行质粒复制。
按能否自主转移,可以将天然存在的质粒分为转移型质粒和非转移型质粒两大类。这里要注意的是,获得质粒的细菌可随之而获得一些生物学特性,如耐药性或产生细菌素的能力等。从环境友好出发,实验室里的废弃菌液,一定要灭过菌才能倒哦。
穿梭质粒:是指一类人工构建的具有两种不同复制起点和筛选,因而可以在两种不同类群宿主中存活和复制的质粒载体。此概念不仅用于不同的微生物菌群之间,也可以推广到真核生物表达载体的构建,如用于枯草的pBE2、酵母的pPIC9K、哺乳动物表达载体pMT2和用于植物细胞的Ti质粒这些穿梭质粒不仅可以在大肠杆菌中复制扩增,也可以在相应的枯草、酵母、动物或植物细胞中扩增和表达。这样利于对质粒的分子生物学操作和大量制备。
3.质粒的不相容性
通俗来说,就是一山不能容二虎。但是作为科学来说,我们需要一个严谨的定义:"利用同一复制系统的两个质粒会在复制和随后向自细胞的分配过程中彼此竞争,这样的质粒在细菌培养物中不能和平共处,这种现象称之为不相容性”。

5.2怎么才能在一个菌里面使用两个质粒呢?
简单的方法就是使用不同复制源的且带有不同抗性基因的两个质粒。
5.3构建质粒PCR常见问题
扩增基因的过程并不困难,但未必能保证百分之百的成功率,可能原因:
1.结果出现引物二聚体、非特异性条带(大小不对)的现象,可以通过降低模板和引物的浓度、降低镁离子浓度、适当减少酶量,提高退火温度,来提高扩增特异性。
2.出现凝胶条带弥散状态,多为模板不纯、反应体系中各成分比例不合适、退火温度设置偏低、循环次数过多等原因。
3.长片段基因的扩增容易增加点突变和错配率,选用高扩增能力、高保真、可靠性强的聚合酶也很关键。

5.4无法成功克隆到基因
原因:引物设计不合理,模板质量不高。
解决方法:多找几个模板同时进行克隆;重新评估引物的合理性和特异性,比如将引物长度增至40bp左右。
5.5酶切位点的选择
这一点很重要。通常很多同学在选择酶切位点时,会习惯性的选择师兄师姐们常用的酶切位点,比如BamHI等,但实际上这样是不行的。此时需要注意:目的基因中不能出现所选酶切位点,否则,在连接时,残留的酶会对目的基因进行切割,这将直接导致实验的失败。
解决办法:选择酶切位点前,需要检查所选酶切位点是否存在于目的基因中。
5.6如果构建质粒载体失败了,怎么办?
构建质粒载体不成功。我们可以根据引物设计是否合理?酶切位点是否选择正确?模板质量是否较好?PCR程序是否设置正确?如果以上几点都没问题,那载体构建就不会出问题。
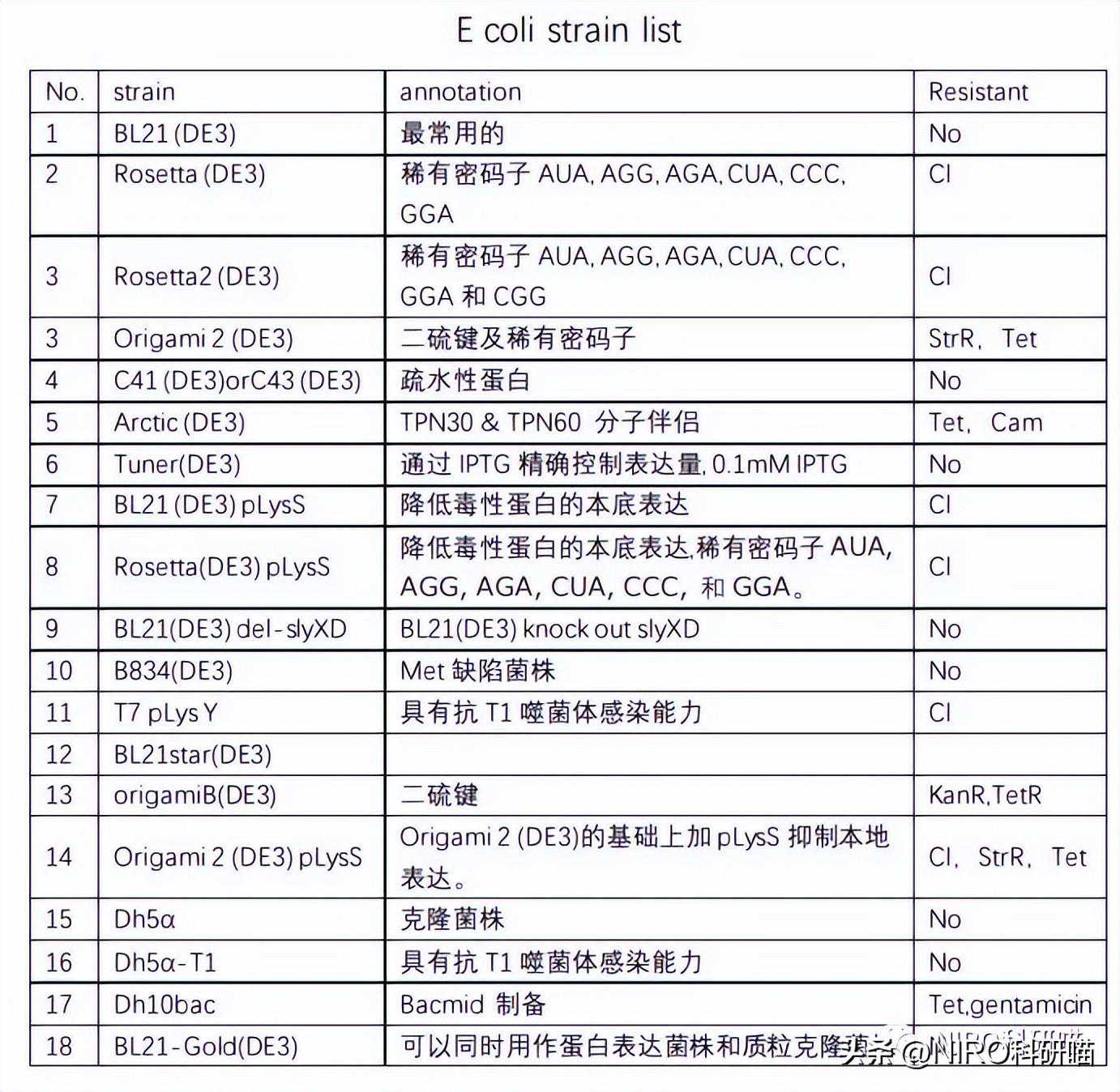
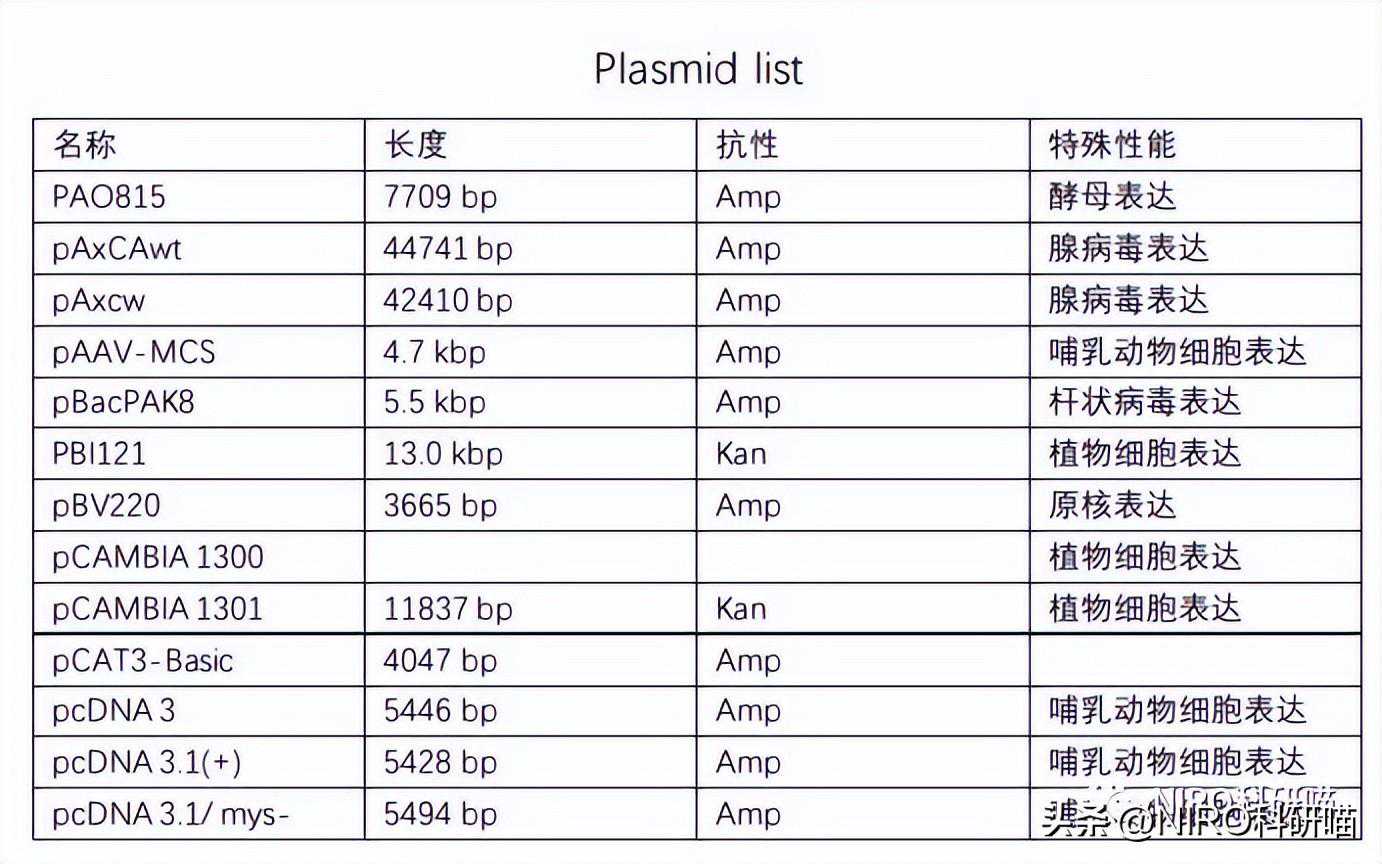
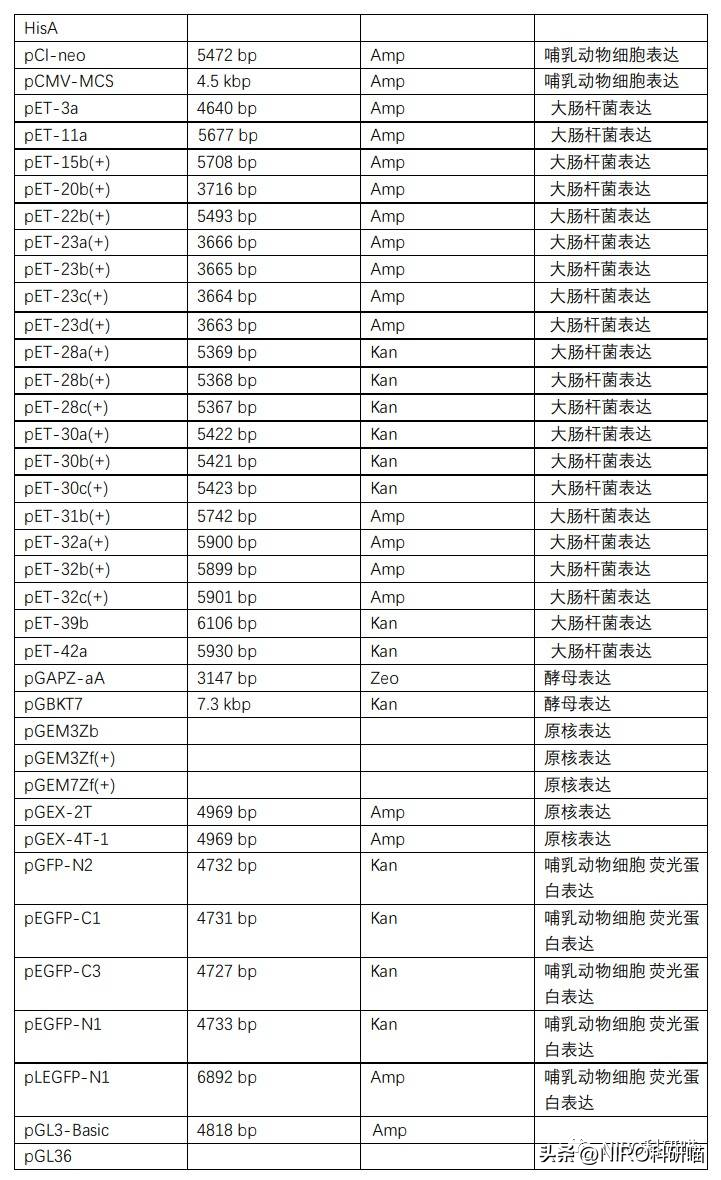
5.7如何选择正确的菌体和载体
5.8其他问题
在操作酶切连接的步骤时也要定性定量,保证酶活性充足,例如:双酶切尽可能选择同种缓冲buffer,一般用量40U单位以上(用量不超过总体积1/10),保证酶切过程充分;可根据目标片段量(ng)=(载体量(ng)x目标片段长度(kb)/(载体DNA片段长度(kb))x目片段和载体的摩尔比(1:3-1:8)计算出目标片段和载体的加入量,提高连接效率。
构建好的载体放入感受态细胞中转化(TOP10、DH5a、BL21等,感受态细胞在使用时尽可能保证新鲜,避免反复冻融,冰上孵育和热激时间应严格控制),经抗性、菌落PCR、酶切、测序等多道程序验证,确定是否转化成功,最终获得正确的重组克隆基因片段。

扫
码
关
注
NIRO科研喵
点个在看分享每日新知识
本文链接:https://nyjx.s9479.cn/509454492056.html
上一篇:立秋过后采收忙
立秋过后采收忙
下一篇:锰矿的选矿方法
锰矿的选矿方法
